Oversetting i bakterier
Med fokus på E. coli
© Anders Skovly 2025
Denne artikkelen handler om hvordan basesekvenser i RNA oversettes til aminosyresekvenser i proteiner.
mRNA, tRNA, og ribosomer
I artikkelen «proteiner og nukleinsyrer» ble det skrevet at "Et gen er en basesekvens i DNA som kan oversettes til aminosyresekvensen i et protein. Oversettingen av et gen til et protein er indirekte: først blir basesekvensen i DNA transkriptert ("kopiert") til RNA, og deretter blir basesekvensen i RNA oversatt til aminosyresekvensen i et protein."
Dette var en forenkling da det også finnes gener som transkripteres til RNA, men som ikke oversettes til en aminosyresekvens. Gener som oversettes til proteiner kalles protein-kodende gener, mens gener som ikke oversettes til protein kalles RNA-kodende gener. De RNA som oversettes til aminosyresekvenser kalles messenger-RNA (mRNA), mens de RNA som ikke kan oversettes har ulike navn ut ifra hva de gjør i cellen. To eksempler er ribosomale RNA (rRNA) og transfer-RNA (tRNA), som begge er viktige komponenter i oversettingsprosessen.
Et ribosom er en stor molekylstruktur bestående av flere ulike proteiner og ribosomale RNA som holdes sammen av ikke-kovalente bindinger. Ribosomers rolle i oversetting ligner på RNA polymerase sin rolle i transkripsjon: Et ribosom binder til mRNA, sørger for at to «komplementære» aminosyrer binder til mRNA, og katalyserer dannelsen av et kovalent bånd mellom aminosyrene. Deretter beveger ribosomet seg langs RNA mens nye «komplementære» aminosyrer binder mRNA, og kovalente bånd dannes mellom aminosyrene slik at det dannes en aminosyrekjede, altså et protein.
Aminosyrer binder ikke direkte til mRNA. I stedet binder hver aminosyre til en tRNA, og tRNA binder deretter til mRNA. Altså er det en indirekte binding mellom aminosyren og mRNA.
Denne figuren viser strukturen til en tRNA (figuren viser en tRNA fra gjær, tRNA i bakterier har generelt liknende struktur). En tRNA har fire intra-molekylære baseparede regioner, altså fire sett komplementære sekvenser innad i samme RNA som baseparer med hverandre. Slike regioner kalles ofte for "stammer". På enden av tre av stammene finnes tre "løkker", mens den fjerde stammen ender i tRNA's 3'- og 5'-ender.
Stammene og løkkene foldes på en slik måte at tRNA får en 3D-struktur som ligner på en L. tRNA's 3'-ende er da posisjonert på den ene enden av L-en. En aminosyre kan binde til en tRNA ved at det dannes et kovalent bånd mellom aminosyren's karboksylsyre og tRNA's 3'-hydroksyl.
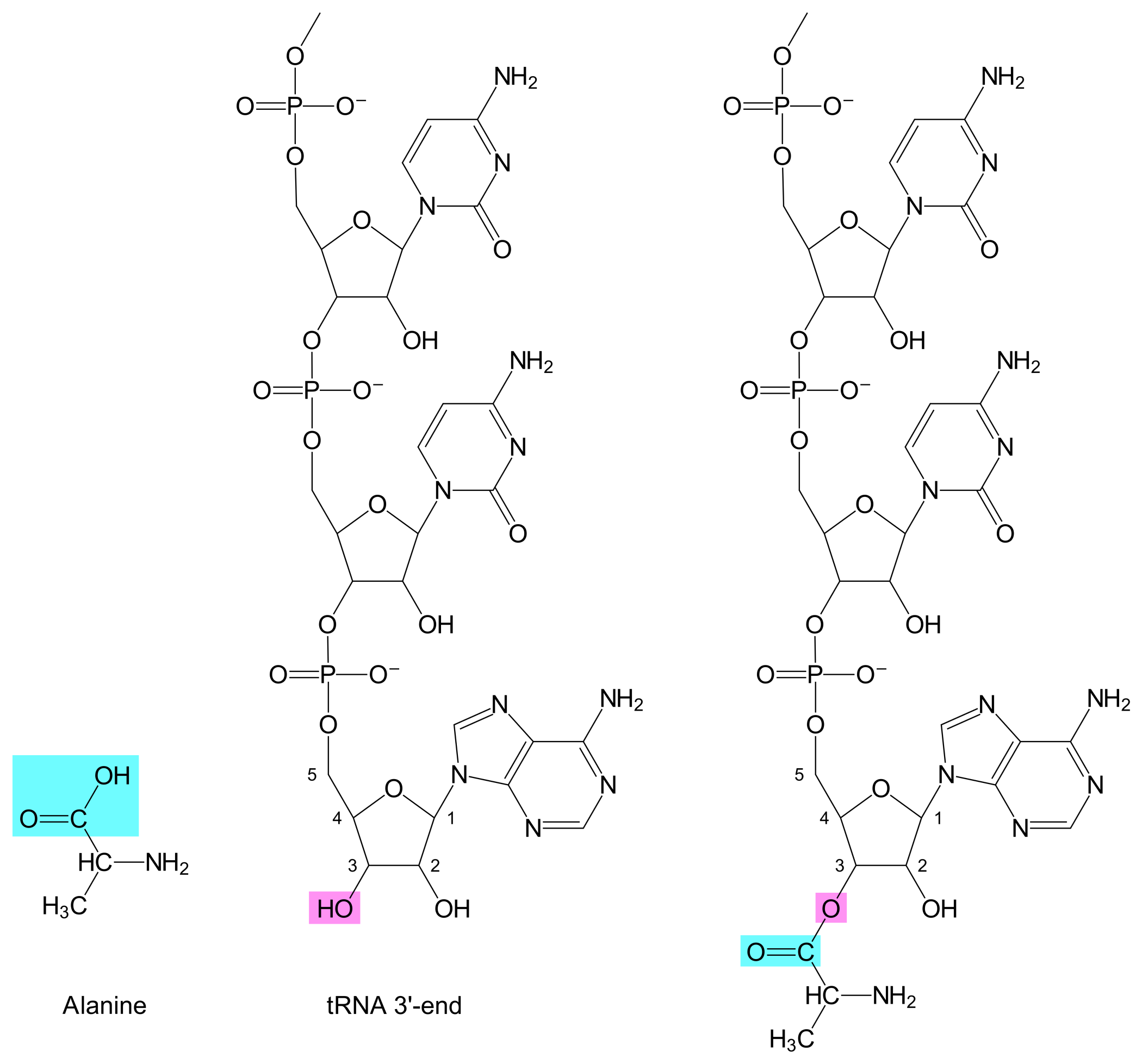
En tRNA kan binde til en mRNA via baseparing mellom en base-triplett i mRNA og en basetriplett i tRNA. Basetripletten i mRNA kalles en kodon, mens basetripletten i tRNA kalles en antikodon. Antikodonet befinner seg i en av de tre tRNA-løkkene, og er posisjonert på motsatt side av L-en i forhold til 3'-hydroksylen (se figuren linket ovenfor).
Oversettelse av lacZ
Oversetting beskrives her med et praktisk eksempel: oversetting av lacZ-meldingen i mRNA til et LacZ-protein. Som nevnt i artikkelen om laktose-promoteren så blir genene lacZ, lacY, og lacA transkriptert til tre protein-meldinger i en og samme mRNA. Til tross for dette så oversettes de tre meldingene hver for seg. Sekvensen i lacZ-meldingen er presentert her i form av nontemplat-sekvensen til lacZ-genet, vist med rød tekst.
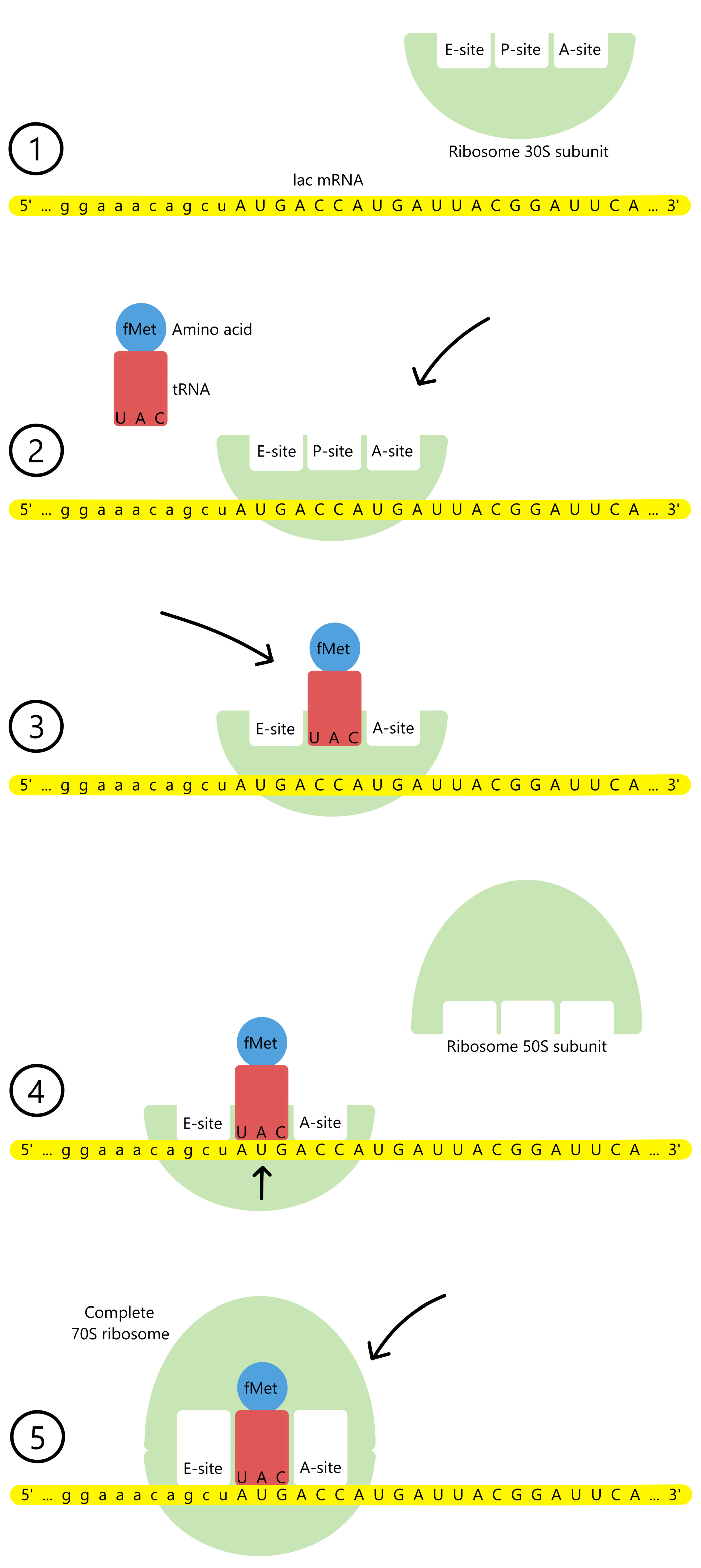
Før oversettingen av lacZ-meldingen starter eksisterer ribosomet i to deler: en liten ribosom-del (30S-del) og en stor ribosom-del (50S-del). (Den lille delen har en sedimentasjonskoeffisient på 30 Svedberg-enheter og den store delen har en sedimentasjonskoeffisient på 50 Svedberg-enheter, derav betegnelsene 30S og 50S. Sedimentasjonskoeffisienter har ingenting med oversetting å gjøre, så det er ikke nødvendig å forstå hva dette er for noe.)
Oversettingen begynner ved at ribosomets 30S-del binder til mRNA, ved 5’-enden av lacZ-meldingen. Deretter binder 30S-delen også til en tRNAfMetUAC. Denne tRNA har antikodon UAC og er bundet til aminosyren N-formyl-Methionin (forkortet fMet). Se figuren nedenfor. (Alternativt kan oversettingen begynne med at 30S-delen først binder til tRNAfMetUAC og deretter til mRNA, disse to hendelsene trenger ikke å skje i noen bestemt rekkefølge.)
(Basesekvenser skrives konvensjonelt fra 5’-enden mot 3’-enden, men i denne artikkelen vil antikodon-sekvenser skrives fra 3’-enden mot 5’-enden ettersom antikodoner må presenteres slik i figurene. Alle kodon-sekvenser er skrevet på den konvensjonelle måten, fra 5'-enden mot 3'-enden.)
Deretter binder tRNA’s UAC-antikodon til en AUG-kodon i mRNA, slik at hver av de tre komponentene i komplekset (mRNA, tRNA, 30S-del) er bundet til begge de to andre. AUG-kodonet fungerer som lacZ-meldingens start-kodon. Ribosomets 50S-del binder så til komplekset, og med dette dannes et komplett 70S-ribosom som er klart til å oversette lacZ-meldingen.
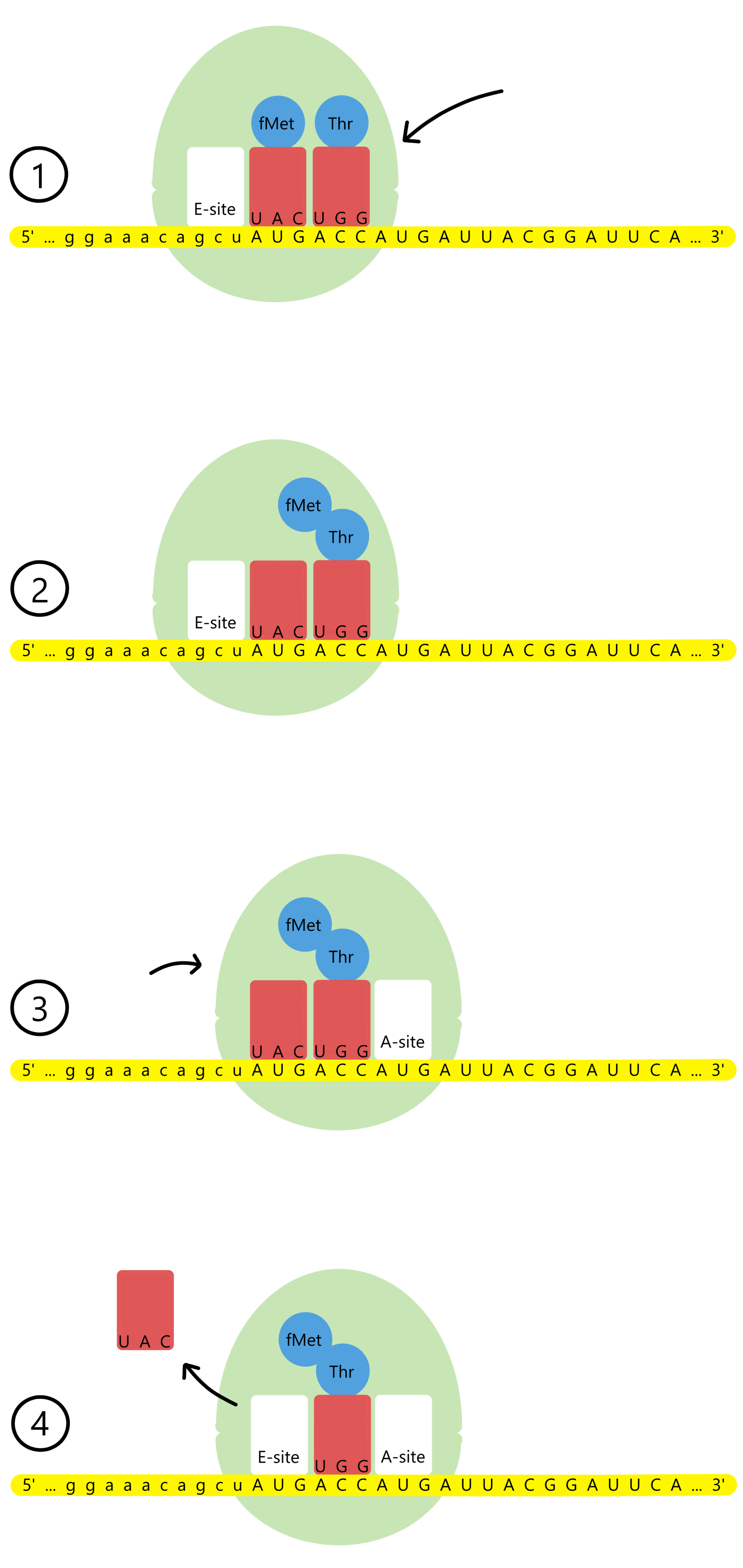
Ribosomet har et spor som mRNA kan bevege seg gjennom, og langs sporet finnes tre bindesteder for tRNA: A-stedet, P-stedet, og E-stedet (A for aminoacyl, P for peptidyl, og E for exit). P-stedet fylles på dette øyeblikket av tRNAfMetUAC baseparet til start-kodonet AUG. Samtidig er lacZ-meldingens andre kodon, ACC, eksponert i A-stedet.
En tRNAThrUGG med aminosyren Threonin (forkortet Thr) entrer A-stedet og baseparer til andre kodon. Ribosomet kan nå katalysere dannelsen av et kovalent bånd mellom aminosyrene fMet og Thr. Samtidig brytes det kovalente båndet mellom fMet og tRNAfMetUAC. Resultatet er at tRNAThrUGG nå er bundet til en Thr som videre er bundet til en fMet.
Etter bånddannelsen beveger ribosomet seg en distanse på tre nukleotider langs mRNA, i nedstrøms retning (mot mRNA’s 3’-ende). Dette gjør at start-kodonet samt tRNAfMetUAC flyttes fra P-stedet til E-stedet, andre kodon samt tRNAThrUGG flyttes fra A-stedet til P-stedet, og lacZ-meldingens tredje kodon, enda en AUG, blir eksponert i A-stedet. tRNAfMetUAC løsner fra E-stedet.
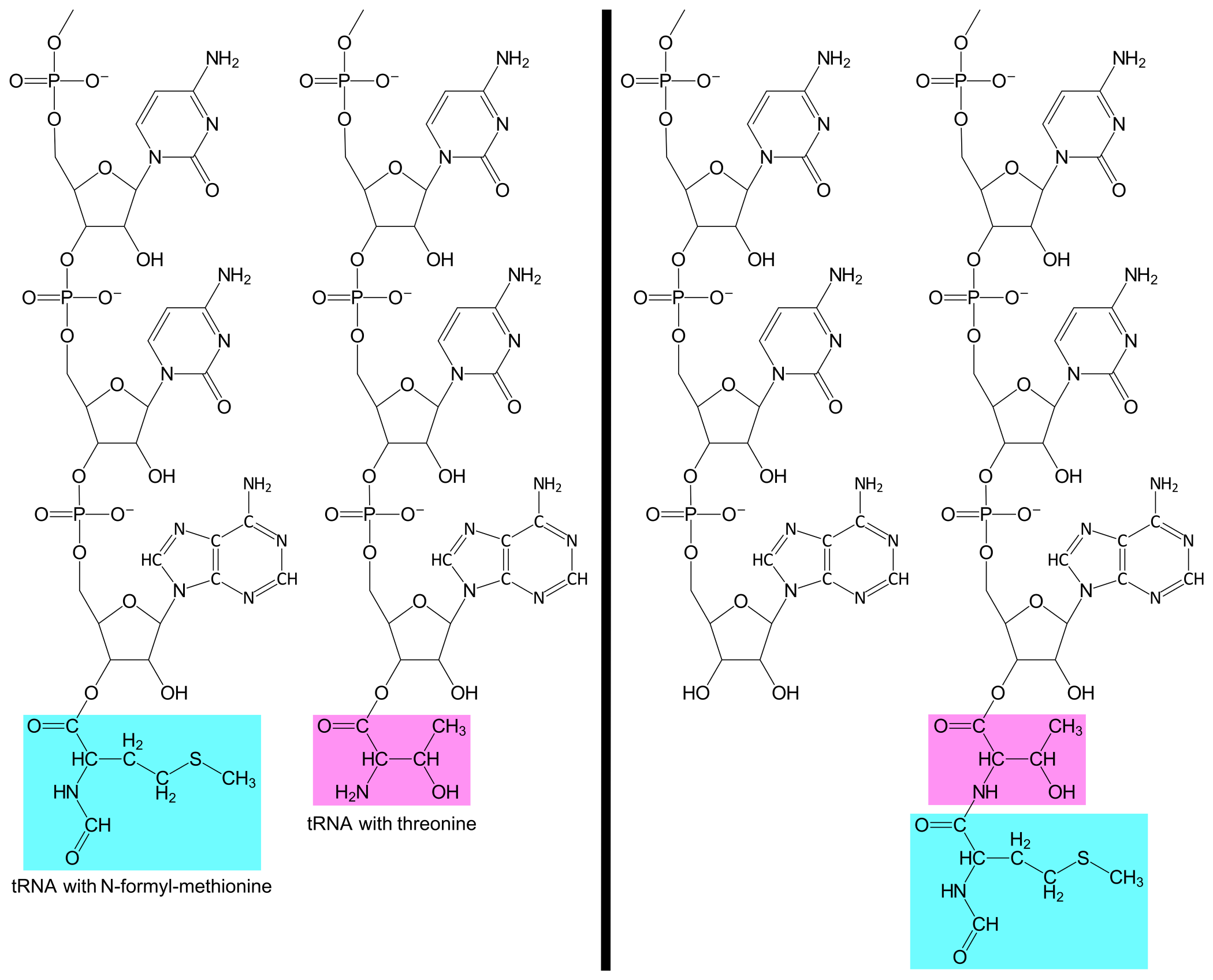
En tRNAMetUAC med en Methionin entrer A-stedet og baseparer til tredje kodon, AUG. Ribosomet katalyserer dannelsen av et kovalent bånd mellom Thr og Met. Samtidig brytes båndet mellom Thr og tRNAThrUGG. Resultatet er at tRNAMetUAC nå er bundet til en Met, som videre er bundet til en Thr, som videre er bundet til en fMet.
Ribosomet beveger seg igjen en distanse på tre nukleotider nedstrøms langs mRNA, slik at andre kodon samt tRNAThrUGG flyttes fra P-stedet til E-stedet, tredje kodon samt tRNAMetUAC flyttes fra A-stedet til P-stedet, og lacZ-meldingens fjerde kodon, AUU, blir eksponert i A-stedet. tRNAThrUGG løsner fra E-stedet.
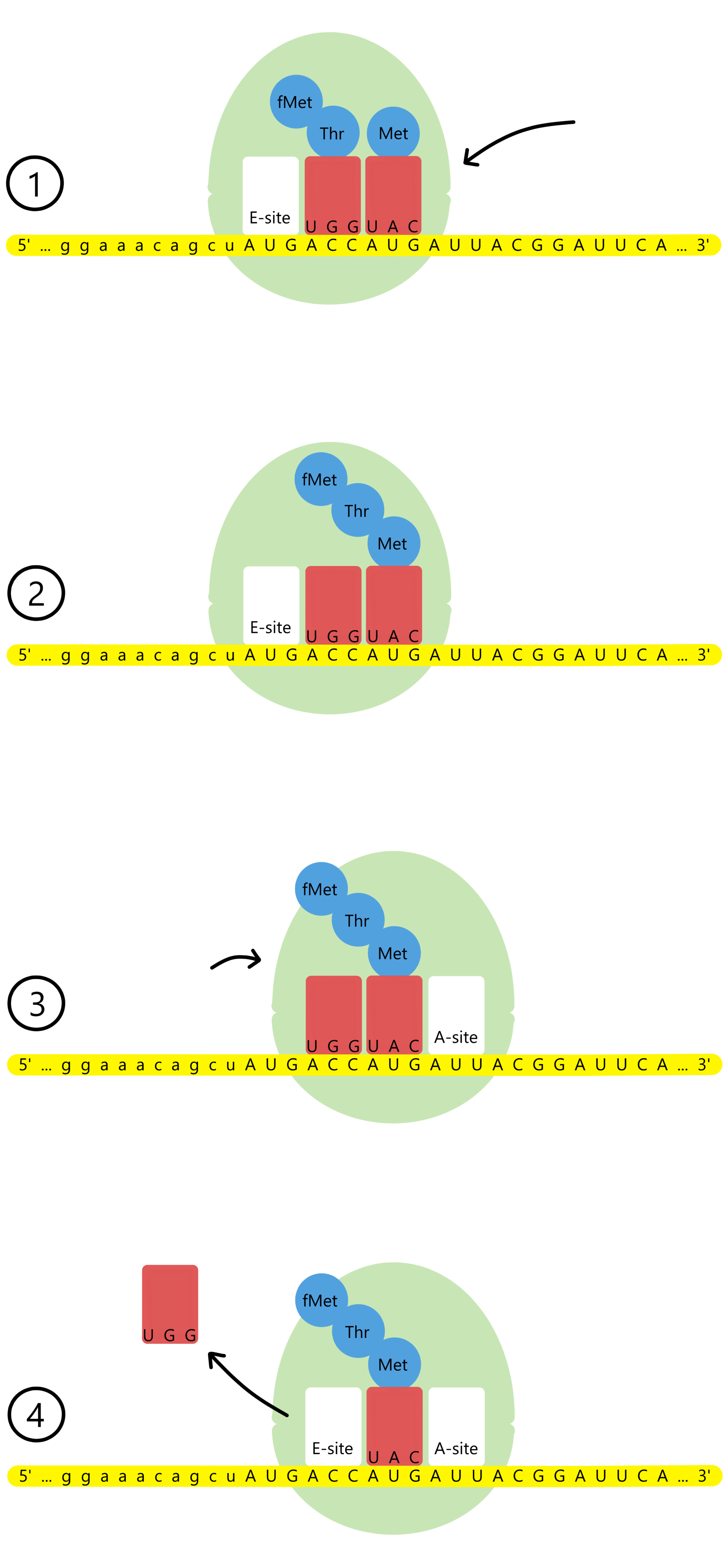
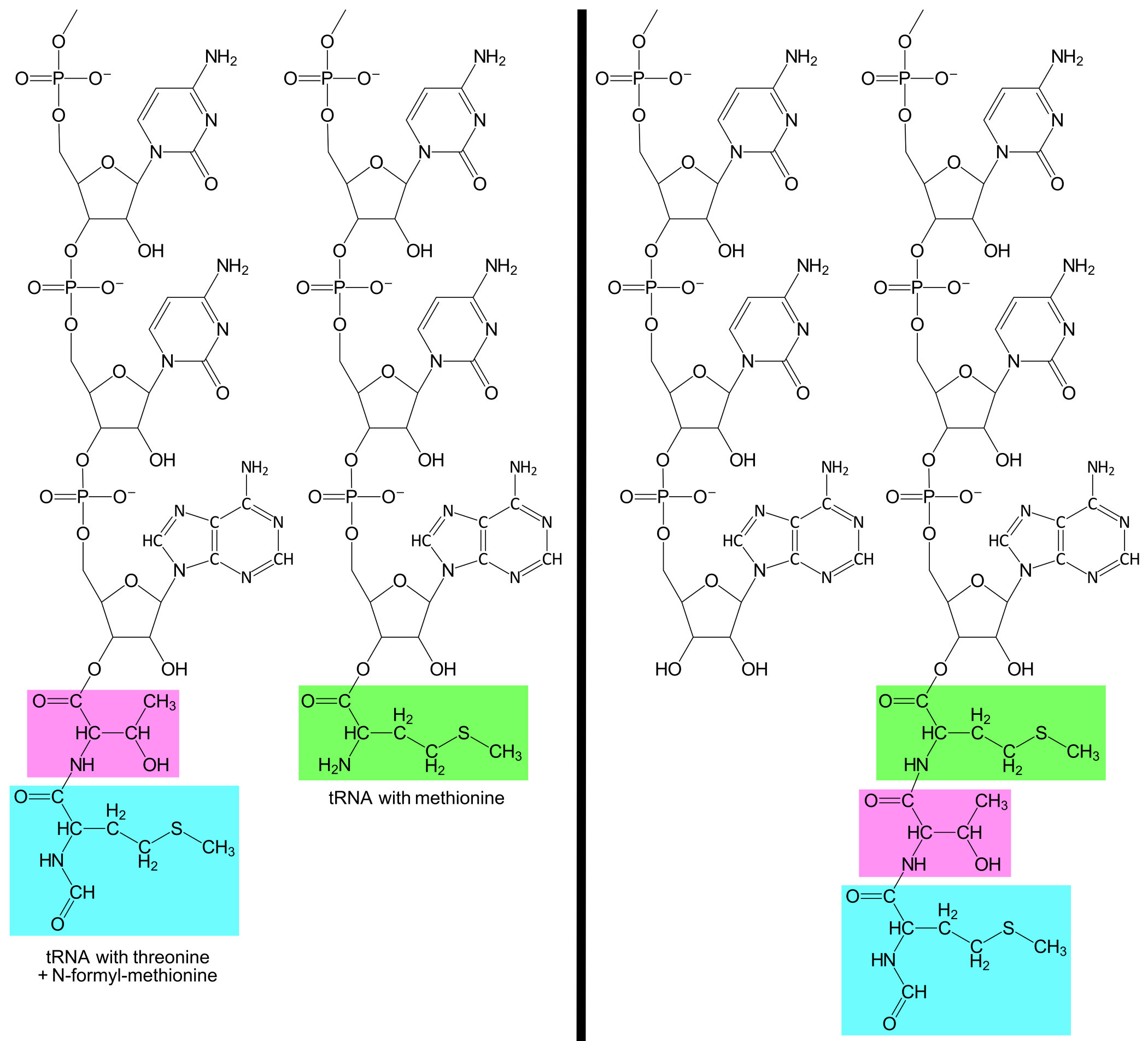
Oversettingen fortsetter med gjentakende sykluser av binding av tRNA til A-stedet, dannelse av kovalent bånd mellom aminosyrer, flytting av ribosomet langs mRNA, og løsning av tRNA fra E-stedet. Med dette vokser aminosyrekjeden lengre og lengre.
Til slutt vil aminosyrekjeden ha vokst til å inneholde 1024 aminosyrer og danner da et komplett LacZ-protein (β-galaktosidase). lacZ-meldingens siste kodon, UAA, blir eksponert i A-stedet. UAA skiller seg fra alle andre kodoner i lacZ-meldingen i at det ikke binder til en tRNA. I stedet vil et protein kalt Release Factor 1 (RF1) entre A-stedet og binde UAA.
RF1 katalyserer dannelsen av et kovalent bånd mellom et vannmolekyl og siste aminosyre’s karboksylsyre. Samtidig vil det kovalente båndet mellom aminosyren og tRNA’s 3’-oksygen brytes. Dette frigjør LacZ-proteinet fra tRNA og fra ribosomet. Etter dette løsner ribosomets 30S- og 50S-deler samt mRNA, tRNA og RF1 fra hverandre, og oversettingen er ferdig.
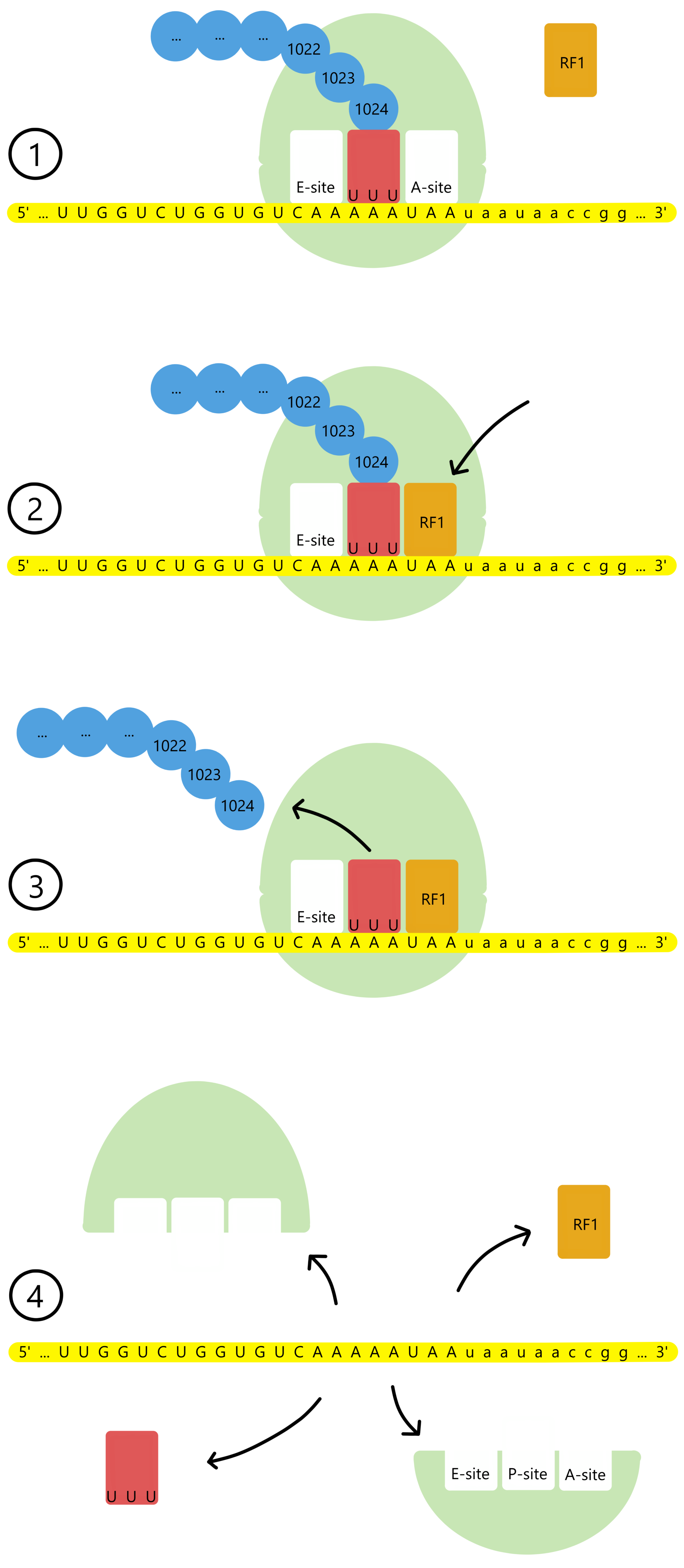
Oversetting generelt
Ut i fra eksempelet med oversetting av lacZ-meldingen kan den generelle oversettingsprosessen beskrives slik:
Et gen, samt protein-meldingen som transkripteres fra genet, består av en rekke av basetripletter kalt kodoner. Et ribosom kan binde til mRNA ved starten av protein-meldingen. Deretter vil ribosomet oversette en og en av meldingens kodoner til aminosyrer. Først oversettes start-kodonet, så oversettes andre kodon, så tredje kodon, og så videre. Dette krever at ribosomet beveger seg langs meldingen, i retning mot mRNA's 3'-ende.
Oversettingen utføres ved hjelp av tRNA bundet til aminosyrer. tRNA's antikodoner baseparer med meldingens kodoner. For hvert par av nabo-kodoner vil ribosomet katalysere dannelsen av et kovalent bånd mellom de to oversatte nabo-aminosyrene. Først blir første og andre aminosyre bundet sammen. Deretter blir andre og tredje aminosyre bundet sammen. Deretter blir tredje og fjerde aminosyre bundet sammen, og så videre. Denne gjentakende prosessen skaper en kjede av aminosyrer.
Oversettingen fortsetter frem til ribosomet møter meldingens stopp-kodon. En release-factor binder stopp-kodonet, og katalyserer så brytingen av det kovalente båndet mellom siste aminosyre og siste tRNA. Med dette frigjøres kjeden av aminosyrer fra ribosomet, og oversettingen avsluttes.
Om kodoner og antikodoner
Ettersom hvert kodon inneholder tre baser, og ettersom hver base kan være en av fire forskjellige (A, C, G, eller U), så finnes det 4 × 4 × 4 = 64 ulike kodoner. Kodon UAG kan bindes av Release Factor 1 (RF1), kodon UGA kan binde RF2, og kodon UAA kan binde enten RF1 eller RF2. Begge release-factorene har samme funksjon: de frigjør aminosyrekjeden fra tRNA, slik at oversettingen av en protein-melding avsluttes. De tre kodonene UAG, UGA, og UAA kalles derfor for stopp-kodoner.
De andre 61 kodonene oversettes til aminosyrer. I E. coli finnes det dog ikke 61 ulike tRNA/antikodoner, det finnes kun 41 ulike. Videre så er det kun 21 ulike aminosyrer. Altså må noen kodoner binde samme tRNA/antikodon, og noen tRNA må binde samme aminosyre. For eksempel, kodoner GCA og GCG binder begge til tRNAAlaCGU, mens kodoner GCC og GCU begge binder til tRNAAlaCGG. Altså fire ulike kodoner som binder to ulike tRNA som har en og samme aminosyre, Alanin.
| Kodon | tRNA/RF | Kodon | tRNA/RF |
|---|---|---|---|
| AAA | tRNALysUUU | CAA | tRNAGlnGUU |
| AAG | tRNALysUUU | CAG | tRNAGlnGUU, tRNAGlnGUC |
| AAC | tRNAAsnUUG | CAC | tRNAHisGUG |
| AAU | tRNAAsnUUG | CAU | tRNAHisGUG |
| ACA | tRNAThrUGU | CCA | tRNAProGGU |
| ACG | tRNAThrUGU, tRNAThrUGC | CCG | tRNAProGGU, tRNAProGGC |
| ACC | tRNAThrUGG | CCC | tRNAProGGG |
| ACU | tRNAThrUGG | CCU | tRNAProGGG |
| AGA | tRNAArgUCU | CGA | tRNAArgGCI |
| AGG | tRNAArgUCU, tRNAArgUCC | CGG | tRNAArgGCC |
| AGC | tRNASerUCG | CGC | tRNAArgGCI |
| AGU | tRNASerUCG | CGU | tRNAArgGCI |
| AUA | tRNAIleUAL | CUA | tRNALeuGAU |
| AUG | tRNAMetUAC | CUG | tRNALeuGAU, tRNALeuGAC |
| AUC | tRNAIleUAG | CUC | tRNALeuGAG |
| AUU | tRNAIleUAG | CUU | tRNALeuGAG |
| GAA | tRNAGluCUU | UAA | RF1, RF2 |
| GAG | tRNAGluCUU | UAG | RF1 |
| GAC | tRNAAspCUG | UAC | tRNATyrAUG |
| GAU | tRNAAspCUG | UAU | tRNATyrAUG |
| GCA | tRNAAlaCGU | UCA | tRNASerAGU |
| GCG | tRNAAlaCGU | UCG | tRNASerAGU, tRNASerAGC |
| GCC | tRNAAlaCGG | UCC | tRNASerAGG |
| GCU | tRNAAlaCGG | UCU | tRNASerAGG |
| GGA | tRNAGlyCCU | UGA | RF2 (tRNASecACU) |
| GGG | tRNAGlyCCU, tRNAGlyCCC | UGG | tRNATrpACC |
| GGC | tRNAGlyCCG | UGC | tRNACysACG |
| GGU | tRNAGlyCCG | UGU | tRNACysACG |
| GUA | tRNAValCAU | UUA | tRNALeuAAU |
| GUG | tRNAValCAU | UUG | tRNALeuAAU, tRNALeuAAC |
| GUC | tRNAValCAG | UUC | tRNAPheAAG |
| GUU | tRNAValCAG | UUU | tRNAPheAAG |
Det at noen antikodoner kan binde to ulike kodoner er mulig fordi bindingen mellom kodon og antikodon ikke eksklusivt er basert på AU-par og CG-par. Dersom kodonets tredje base (altså basen ved kodonets 3’-ende) er en G, så kan denne G basepare med enten C eller U i antikodonet. Og dersom kodonets tredje base er U, så kan denne U basepare med enten A eller G i antikodonet. Altså kan det dannes GU-par mellom kodonet og antikodonet, men kun med kodonets tredje base. Et eksempel på dette er kodon GGG, som kan binde både tRNAGlyCCU og tRNAGlyCCC.
Binding mellom kodon og antikodon vil i noen tilfeller involvere en av to uvanlige baser kalt inosin og lysidin (forkortet I og L). Hos E. coli finnes inosin i tRNAArgGCI, hvor antikodonet GCI kan binde de tre kodoner CGA, CGC og CGU. Lysidin finnes i tRNAIleUAL, hvor antikodonet UAL kan binde kodonet AUA.
(For å være nøyaktig: inosin og lysidin er egentlig ikke navn på baser, det er navn på nukleosider. (En nukleosid er en base bundet til en (deoksy)ribose, i kontrast til det svært like ordet nukleotid, som refererer til en base bundet til en (deoksy)ribose bundet til en eller flere fosfater.) Forskjellen mellom baser og nukleosider er ikke viktig her, så for enkelhets skyld vil jeg kalle inosin og lysidin for baser.)
Oversettingsfaktorer
De ulike fasene i oversettingsprosessen, altså sammensetting av komplett ribosom på mRNA, dannelse av kovalente bånd mellom aminosyrer, og dissosiering av ribosomet fra mRNA og det ferdige proteinet, refereres til som initiering, elongering, og terminering. I tillegg til 30S-delen, 50S-delen, mRNA, og tRNA, så involverer oversetting også en rekke proteiner. Disse er Initieringsfaktorer 1, 2, og 3 (IF1, IF2, og IF3), elongeringsfaktorer Tu, Ts, og G (EF-Tu, EF-Ts, og EF-G), release-faktorer 1, 2, og 3 (RF1, RF2, RF3), samt ribosom resirkulering-faktor (RRF). Funksjonen til RF1 og RF2 har blitt beskrevet. Funksjonen til de andre faktorene vil ikke dekkes i denne artikkelen.
Initiator-tRNA og start-kodoner
I eksempelet med oversetting av lacZ-meldingen så bindes start-kodonet AUG av en tRNAfMetUAC. Tredje kodon, som også er en AUG, bindes istedet av en tRNAMetUAC. Førstnevnte tRNA kalles en initiator-tRNA mens sistnevnte tRNA kalles en elongator-tRNA. Av alle de 41 ulike antikodoner i E. coli, så er det kun antikodon UAC som finnes både på en initiator-tRNA og på en elongator-tRNA. Hver av de resterende 40 antikodoner finnes kun på en elongator-tRNA. E. coli har derfor 41 ulike elongator-tRNA samt én eneste initiator-tRNA, altså initiator-tRNAfMetUAC.
Initiator-tRNA er den eneste tRNA som kan binde til P-stedet i ribosomets 30S-del før 30S-delen og 50S-delen er satt sammen til et komplett 70S-ribosom. Dette betyr at oversetting alltid starter med en initiator-tRNAfMetUAC, uavhengig av hvilken protein-melding som oversettes. Initiator-tRNA kan derimot ikke binde til A-stedet i 70S-ribosomet, det er det kun elongator-tRNA som kan gjøre. Dette kommer av at initiator-tRNA og elongator-tRNA har forskjellige molekylstrukturer.
(fMet er en modifisert Methionin hvor nitrogen-atomet i aminen er kovalent bundet til en formyl, derav navnet N-formyl-Methionin. Jeg vet ikke nok om denne formyl-modifikasjonen til å kommentere på dens funksjon. I eukaryotiske organismer har initiator-tRNA en vanlig Methionin istedet for en N-formyl-Methionin, slik at initiator-tRNAMetUAC og elongator-tRNAMetUAC begge har samme aminosyre.)
Hvis oversetting alltid starter med en initiator-tRNAfMetUAC, betyr dette at alle gener i E. coli starter med kodonet AUG? Nei, faktisk ikke. I visse gener benyttes enten GUG, UUG, eller AUU som start-kodon. Uavhengig av hvilken basetriplett som fungerer som start-kodon så vil det være initiator-tRNAfMetUAC som binder til start-kodonet.
(Jeg er ikke helt sikker, men i tilfellene med GUG eller UUG som start-kodon så tror jeg ikke at første base i kodonet danner et basepar med antikodonet i initoator-tRNA. Og i tilfellet med AUU så tror jeg ikke at tredje base i kodonet danner basepar med antikodonet. Altså, jeg tror ikke det kan dannes GU-par eller UU-par som involverer kodonets første base, eller UU-par som involverer kodonets tredje base. Istedet tror jeg at initiator-tRNAfMetUAC binder til de alternative start-kodonene via kun tó basepar, som da vil være to vanlige AU- og CG-par.)
(Da hele E. coli's DNA hadde blitt sekvensert ble det funnet totalt 4288 protein-kodende gener (Blattner 1997). Disse 4288 inkluderte beviste gener såvel som foreslåtte gener (DNA-sekvenser som antas å være gener). Av de 4288 genene var det 3542 gener (83%) som hadde AUG som start-kodon, 612 (14%) som hadde GUG som start-kodon, og 130 (3%) som hadde UUG som start-kodon. Senere ble det også identifisert to gener i E. coli som har AUU som start-kodon (Binns 2002). I BioCyc sin database for E. coli, oppdatert i 2023, er det listet 4312 protein-kodende gener. Det er derfor mulig at Blattner's start-kodon-frekvensene er ganske nøyaktige, gitt at de fleste av Blattner's foreslåtte gener er ekte gener og at Blattner stort sett identifiserte korrekt start-kodon.)
tRNASecACU og Selenocysteine-innsettingssekvensen
UGA-tripletter fungerer oftest som stopp-kodoner, men kan i visse tilfeller fungere som selenocystein-kodoner. Dersom UGA er ment å oversettes til selenocystein vil kodonet etterfølges nedstrøms av en SECIS (Selenocysteine-innsettingssekvens, insertion sequence). tRNASecACU binder først sammen med proteinet SelB, og deretter binder SelB til SECIS. tRNASecACU holdes da i posisjon ved UGA-kodonet, og når et ribosom nærmer seg og UGA-kodonet entrer ribosomets A-sted kan tRNASecACU settes inn i A-stedet (se Figur 9 i Heider 1992).
Dersom tRNASecACU ikke binder til A-stedet på denne måten vil et RF2-protein istedet binde til UGA i A-stedet, slik at oversettingen avsluttes. (Flere detaljer om SECIS er utdypt i en egen artikkel.)
Shine-Dalgarno-sekvensen
I E. coli vil majoriteten av protein-meldingene ha en såkalt Shine-Dalgarno-sekvens (SD-sekvens) et kort stykke oppstrøms av start-kodonet. Ribosomet's 30S-del består av ulike ribosomale proteiner samt en 16S ribosomal RNA (rRNA), og SD-sekvensen kan basepare til en komplementær sekvens i 3'-enden av 16S rRNA. For eksempel, syv baser oppstrøms av lacZ-meldingens start-kodon finnes sekvensen AGGA, som kan basepare til UCCU i 3'-enden av 16S rRNA:
||||
16S rRNA: 3'OH-AUUCCUCCACUAG-5’
Oversetting av en protein-melding initieres ved at mRNA og initiator-tRNA begge binder til ribosomets 30S-del. Deretter må start-kodonet i mRNA og antikodonet i initiator-tRNA basepare sammen. Funksjonen til SD-sekvensen er å hjelpe med å posisjonere start-kodonet ved ribosomets P-sted, hvor initiator-tRNA er bundet, slik at start-kodonet og antikodonet lettere kan binde sammen. (Flere detaljer om SD-sekvensen er utdypt i en egen artikkel.)
Hvordan startstedet for oversetting bestemmes
(For de som ikke er kjent med konseptet fri energi vil denne seksjonen være vanskeligere å forstå enn de andre seksjonene i denne artikkelen.)
Oversetting starter med at ribosomets 30S-del binder ved started av en protein-melding i mRNA, og en start-kodon (som oftest en AUG) baseparer med en initiator-tRNA. Hvordan kan 30S-delen skille starten av en protein-melding fra andre steder i mRNA?
Selve start-kodonet, enten det er AUG eller noe annet, kan ikke fortelle ribosomet hvor oversettingen skal starte. Det finnes mange AUG-tripletter som ikke fungerer som start-kodoner, dette kan for eksempel ses i basesekvensen til de tre laktose-genene. (Alle AUG-tripletter som ikke går over flere linjer kan markeres ved å søke med ctrl+f "ATG", hvor ATG må brukes fordi linken viser DNA-sekvensen, ikke mRNA-sekvensen. Et søk i lacZ-meldingen gir for eksempel 56 AUG-tripletter.)
Det var lenge tenkt at Shine-Dalgarno-sekvensen var involvert i ribosomets valg av startsted. I Saito 2020 ble det derimot vist at 30S-deler med muterte 16S rRNA fremdeles kunne binde til korrekte startsteder, til tross for at de mutarte 16S rRNA ikke kunne basepare med SD-sekvensen. Altså er ikke SD-sekvensen av betydning for ribosomets valg av startsted.
Noe som derimot virker å være av betydning er intramolekylær baseparing i mRNA. Som nevnt tidligere så kan to komplementære sekvenser i samme RNA basepare sammen til en "stamme". De to sekvensene i stammen forbindes av en løkke-sekvens. Et eksempel på en stamme-løkke i mRNA vises i Figur 1 i de Smit 1990. (de Smit refererer til stamme-løkker med det alternative navnet "hairpin".)
En stamme er ikke en permanent struktur: de to baseparene sekvensene kan brytes fra hverandre og bli "u-foldet" (unfolded). U-foldede komplementære sekvenser kan senere basepare sammen igjen, slik at stammen gjenoppstår. Når en celle inneholder mange mRNA så vil noen av de komplementære sekvensene finnes i form av stamme-løkker, mens resten vil være u-foldet.
Andelen av de komplementære sekvensene som på et hvilket som helst tidspunkt danner stamme-løkker er relatert til stamme-løkkens strukturelle stabilitet. Stabilitet kvantifiseres med "endringen i fri energi" når de u-foldede sekvensene baseparer sammen til en stammeløkke. Endring i fri energi symboliseres med ΔG, hvor lavere (mer negative) verdier av ΔG betyr mer stabile stamme-løkker.
de Smit innførte en rekke mutasjoner i en stamme-løkke som inneholder start-kodonet til en protein-melding (se de Smit's Figur 2). Noen av mutasjonene var beregnet til å redusere stabiliteten til stamme-løkken, mens andre mutasjoner var beregnet til å øke stabiliteten (se de Smit's Tabell 1). Det ble funnet en god korrelasjon mellom økt stamme-løkke-stabilitet og redusert oversettelse av protein-meldingen (altså en god korrelasjon mellom ΔG og "relative expression", se de Smit's Tabell 1 og Figur 4B). Mutanten med den mest stabile stamme-løkken hadde praktisk talt null oversettelse av protein-meldingen (se mutant 14 i Tabell 1).
En mulig forklaring på korrelasjonen mellom stamme-løkke-stabiliteten og oversettelse av protein-meldingen, er at ribosomer bare kan binde til starten av en protein-melding når denne delen av mRNA er u-foldet. Høyere stabilitet av stamme-løkken betyr færre startsteder i u-foldet form, som betyr redusert ribosom-binding og oversetting.
Hvis dette stemmer, så kan det tenkes at stamme-løkke-stabilitet brukes for å skille starten av en protein-melding fra andre steder i mRNA: starten av protein-meldinger kan ha relativt ustabile stamme-løkker, mens andre steder i mRNA kan danne relativt stabile stamme-løkker. Datamaskin-baserte analyser av protein-meldinger i E. coli har indikert at dette stemmer (se Tuller 2010 og Gu 2010, Tuller refererer til intramolekylær baseparing i mRNA som "folding" mens Gu bruker uttrykket "sekundær struktur"). Det virker derfor som at stamme-løkker er en av faktorene som påvirker hvor ribosomet starter oversettingen.
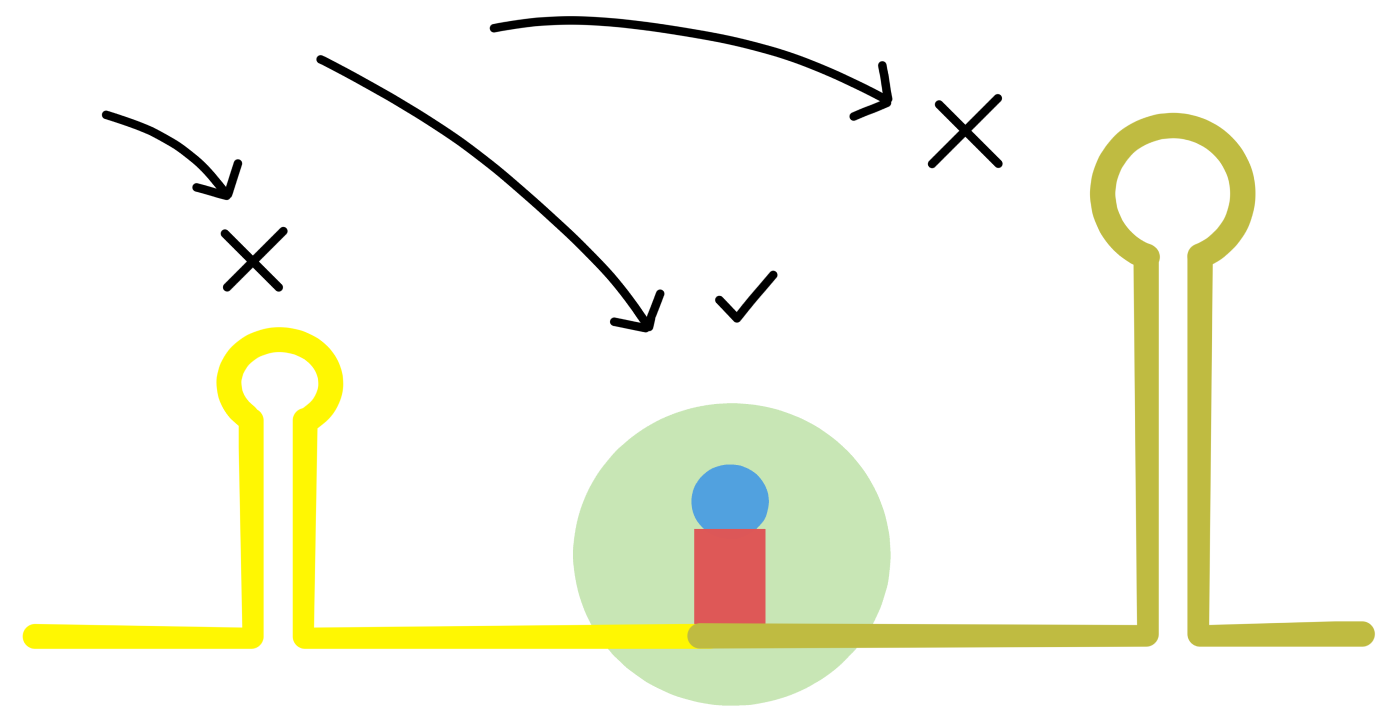
(Man kan spørre: hvis baseparede sekvenser i mRNA kan hindre ribosomet i å binde til mRNA utenfor startstedene for protein-meldingene, kan de ikke også hindre ribosomet i å bevege seg langs mRNA under elongeringsfasen av oversetting? Svaret er at når ribosomet beveger seg langs mRNA så har det evnen til å bryte opp baseparede sekvenser foran seg (Takyar 2005). Denne evnen kalles helikase-aktivitet. Det er likevel godt mulig at stabile baseparede sekvenser kan redusere ribosomets oversettelseshastighet, dette vet jeg ikke.)
Samtidig transkripsjon og oversetting
Under transkripsjon produseres mRNA fra 5’-enden mot 3’-enden, og under oversetting beveger ribosomet seg mot mRNA's 3’-ende. I bakterier kan derfor oversettingen av mRNA starte mens transkripsjonen fremdeles pågår. Faktisk kan et ribosom binde til RNA polymerase før transkripsjon begynner. Ribosomet kan da starte oversetting så snart 5’-enden av mRNA er klar. (Samtidig transkripsjon og oversettelse av en mRNA skjer ikke i eukaryotiske organismer da transkripsjon og oversetting i eukaryoter er fysisk adskilt av kjernemembranen.)
Etter at et ribosom har bundet til starten av en protein-melding og har begynt elongering, så kan et annet ribosom binde til samme melding og begynne å elongere før første ribosom har fullført elongeringen. Derfor kan en melding oversettes av flere ribosomer samtidig, på samme måte som et gen kan transkripteres av flere RNApol samtidig.
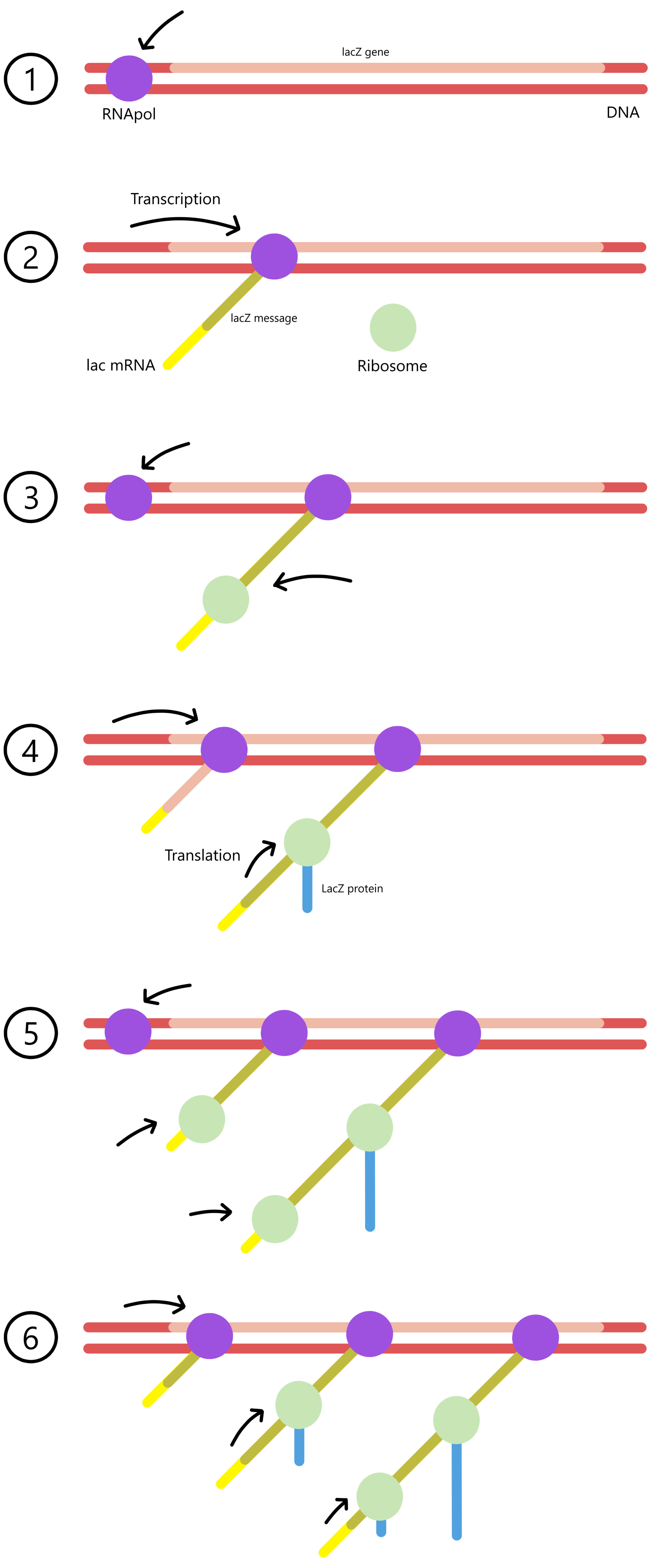
Se samtidig transkripsjon og oversetting i E. coli (trykk på Figur 1 i linken for å åpne bildet). De to tynne strekene som går diagonalt over bildet er DNA. Pilen øverst til venstre peker på det som antakeligvis er en RNA polymerase bundet til promoteren for et ubestemt gen. De svarte kjedene som stikker ut fra DNA er mRNA bundet til ribosomer. I dette bildet transkripteres DNA fra venstre mot høyre, slik at mRNA er korte i venstre del av bildet, og lange i høyre del av bildet. Proteiner er ikke synlige (jeg vet ikke hvorfor). En kort mRNA i høyre del av bildet er muligens produktet av en lengre mRNA som tilfeldigvis har blitt kuttet i to.
Aminoacyl-tRNA synthetaser
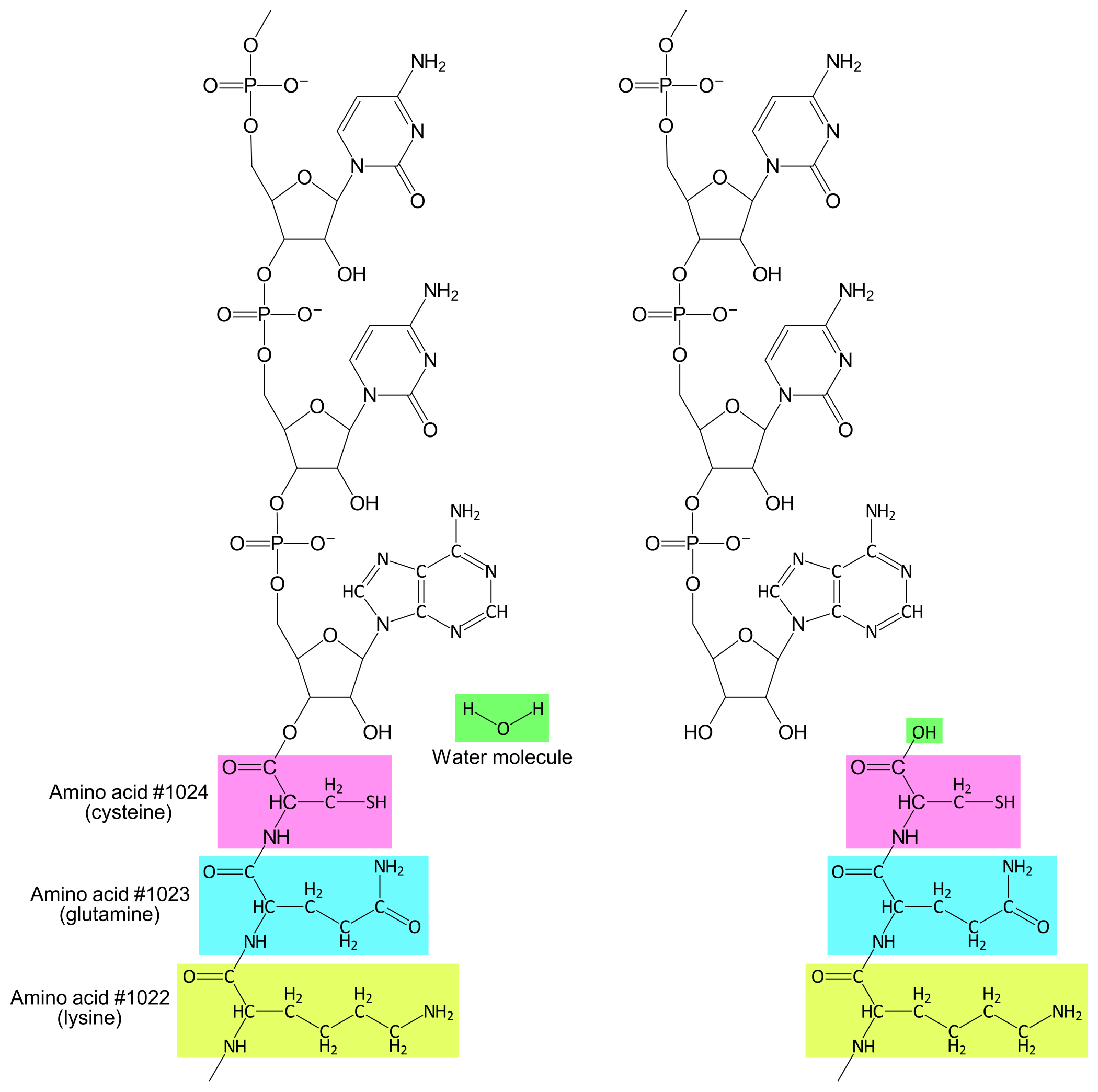
Som nevnt så finnes det 21 ulike aminosyrer og 41 ulike tRNA/antikodoner. For å binde hver aminosyre til den/de riktige tRNA benyttes en samling enzymer kalt Aminoacyl-tRNA synthethaser (AARS). De fleste celler, inkludert E. coli-celler, har tyve ulike AARS som hver kan binde en av de tyve aminosyrene (det finnes ingen AARS for Selenocystein). Hver AARS kan også binde til en eller noen få av de ulike tRNA. En AARS skiller aminosyrer fra hverandre ut ifra molekylstrukturen på den variable gruppen. tRNA skilles også fra hverandre ut ifra basesekvens og molekylstruktur.
Etter at en AARS har bundet både en aminosyre og en tRNA vil AARS katalysere dannelsen av et kovalent bånd mellom 3'-hydroksyl i tRNA og karboksylsyren i aminosyren, slik at det dannes en aminoacyl-tRNA (se Figur 1).
For eksempel, Glutaminyl-tRNA synthetase (GlnRS) er en AARS som kan binde til aminosyren glutamin, og til en av to tRNA: enten tRNAGlnGUC eller tRNAGlnGUU. Når GlnRS binder glutamin og førstnevnte tRNA dannes en Glutaminyl-tRNAGlnGUC. Når GlnRS binder glutamin og sistnevnte tRNA dannes en Glutaminyl-tRNAGlnGUU.
En tRNA som er bundet til en aminosyre sies å være "ladet", mens en tRNA som ikke er bundet til en aminosyre sies å være uladet.
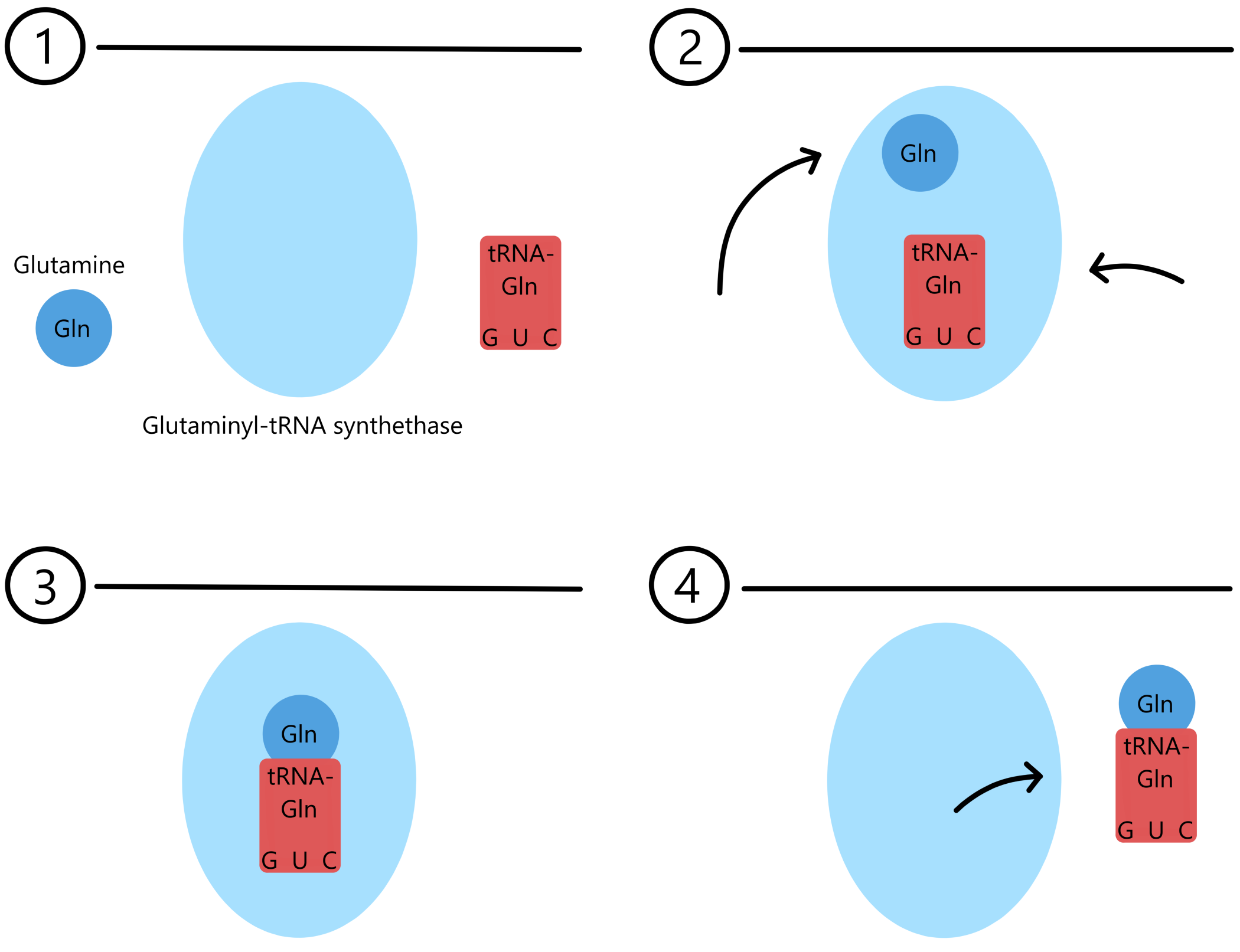
I organismer som benytter Selenocystein (Sec) er dannelse av Selenocysteinyl-tRNASecACU avhengig av enzymene Seryl-tRNA synthetase (SerRS) og Selenocysteine synthetase. Først vil SerRS binde Serin til tRNASecACU og danne Seryl-tRNASecACU, og deretter vil Selenocysteine synthetase modifisere Seryl-tRNASecACU til Selenocysteinyl-tRNASecACU.
("Sec" i tRNASecACU betyr altså at funksjonen til denne tRNA er å oversette en kodon til aminosyren Selenocystein, det betyr ikke at denne tRNA alltid er bundet til Selenocystein.)
Om navnene "messenger-RNA" og "transfer-RNA"
I eukaryoter er transkripsjon og oversetting fysisk adskilt av kjernemembranen. Gener blir transkriptert til mRNA i cellekjernen, mRNA går gjennom kjernemembranen og ut i cytosolen hvor ribosomene befinner seg, og ribosomene oversetter mRNA til proteiner. I eukaryoter er navnet «messenger-RNA» derfor passende. I bakterier, derimot, utføres transkripsjon og oversettelse på samme tid og sted, så navnet messenger-RNA gir egentlig ikke mening i bakterier. Til tross for dette så brukes navnet messenger-RNA i bakterier såvel som i eukaryoter.
Navnet transfer-RNA har opphav i en tekst fra 1958, hvor det ble skrevet at “it was shown that the RNA of a particular fraction of the cytoplasm hitherto uncharacterized became labeled with C14-amino acids in the presence of ATP and the amino acid-activating enzymes, and that this labeled RNA subsequently was able to transfer the amino acid to microsomal protein in the presence of GTP and a nucleoside triphosphate-generating system.” (Hoagland 1958) (Nå som oversettelsesprosessen er bedre forstått ville kanskje «translator-RNA» vært et mer beskrivende navn.)
Oppsummering
Oversettelsen av en basesekvens til en aminosyresekvens involverer basetripletter kalt kodoner i mRNA og antikodoner i tRNA. Oversettelsesprosessen kontrolleres av et ribosom, en stor molekylærstruktur sammensatt fra ulike ribosomale proteiner og ribosomale RNA. Ribosomet katalyserer dannelsen av de kovalente båndene som binder aminosyrer sammen til en kjede.
I tillegg til mRNA, tRNA og ribosom, så er oversetting også avhengig av et sett proteiner kalt initieringsfaktorer, elongeringsfaktorer, release-faktorer og en ribosom-resirkulering-faktor. Oversetting er også avhengig av et sett aminoacyl-tRNA synthethaser som "lader" tRNA med aminosyrer.
I E. coli finnes det én eneste initiator-tRNA, kalt tRNAfMetUAC, samt 41 ulike elongator-tRNA (med 41 ulike antikodoner). Basen på tredje posisjon i et kodon kan danne GU-par med en base i et antikodon, noe som gjør at de 41 ulike tRNA/antikodoner kan binde til 62 ulike kodoner. To av elongator-tRNA har antikodoner med uvanlige baser: en antikodon inneholder basen inosin, en annen antikodon inneholder basen lysidin.
UGA-tripletter fungerer vanligvis som stopp-kodoner, men kan i visse tilfeller fungere som Selenocystein-kodoner. I sistnevnte tilfelle må UGA etterfølges av en SECIS (selenocystein-innsettingssekvens).
Intramolekylær baseparing med høy stabilitet kan hindre binding av ribosom til mRNA. En av faktorene som påvirker hvordan ribosomer binder nær starten av protein-meldinger, og ikke binder til andre steder i mRNA, er at start-stedene generelt ikke danner stamme-strukturer med høy stabilitet, og derfor pleier å være tilgjengelig i u-foldet form.
Et gen kan transkripteres av flere RNApol samtidig, og protein-meldinger i mRNA kan oversettes av flere ribosomer samtidig som at RNApol transkripterer disse mRNA.
Kilder
Binns, Masters
(2002): Expression of the Escherichia coli pcnB gene is translationally limited using an inefficient start codon: a second chromosomal example of translation initiated at AUU. Fri artikkel
Blattner, Ill, Block, Perna, Burland, Riley, Collado-Vides, Glasner, Rode, Mayhew, Gregor, Davis, Kirkpatrick, Goeden, Rose, Mau, Shao
(1997): The Complete Genome Sequence of Escherichia coli K-12. Betalt artikkel
Gu, Zhou, Wilke
(2010): A Universal Trend of Reduced mRNA Stability near the Translation-Initiation Site in Prokaryotes and Eukaryotes. Fri artikkel
Hoagland, Stephenson, Scott, Hecht, Zamecnik
(1958): A soluble ribonucleic acid intermediate in protein synthesis. Fri artikkel
Komine, Adachi, Inokuchi, Ozeki
(1990): Genomic Organization and Physical Mapping of the Transfer RNA Genes in Escherichia coli K12. Betalt artikkel
Saito, Green, Buskirk
(2020): Translational initiation in E. coli occurs at the correct sites genome-wide in the absence of mRNA-rRNA base-pairing. Fri artikkel
de Smit, van Duin
(1990): Secondary structure of the ribosome binding site determines translational efficiency: A quantitative analysis. Fri artikkel
Takyar, Hickerson, Noller
(2005): mRNA Helicase Activity of the Ribosome. Fri artikkel
Tuller, Waldman, Kupiec, Ruppin
(2010): Translation efficiency is determined by both codon bias and folding energy. Fri artikkel