Shine-Dalgarno-sekvensen
© Anders Skovly 2025
Av de ulike faktorene som påvirker initiering av oversettelse i bakterier er det “Shine-Dalgarno-sekvensen” (SD-sekvensen) som har fått mest oppmerksomhet, ihvertfall i biologibøker. I denne artikkelen ser vi på hvordan SD-sekvensen ble oppdaget og hvordan dens rolle i oversetting ble demonstrert. Vi ser også på betydningen av SD-sekvensens lengde og posisjonen.
Initieringsfragmenter
Dersom kopier av en bestemt mRNA blandes med ribosomale 30S- og 50S-deler, initiator-tRNA, og initieringsfaktorer, så vil det dannes komplette 70S-ribosomer på start-stedene til meldingene i mRNA. Så lenge elongator-tRNA og elongeringsfaktorer ikke er til stede i blandingen så kan ikke 70S-ribosomene begynne elongeringen, og vil forbli bundet nær meldingenes start-kodoner.
Ribonukleaser er enzymer som bryter de kovalente båndene som binder sammen ribonukleotidene i en RNA. Hvis ribonukleaser tilsettes blandingen nevnt ovenfor vil det meste av mRNA kuttes opp i biter. RNA-sekvensene nær start-kodonene vil derimot forbli intakte ettersom ribosomer kan beskytte disse sekvensene fra ribonukleasene. De beskyttede sekvensene refereres til som "initieringsfragmenter" ("initiation fragments").
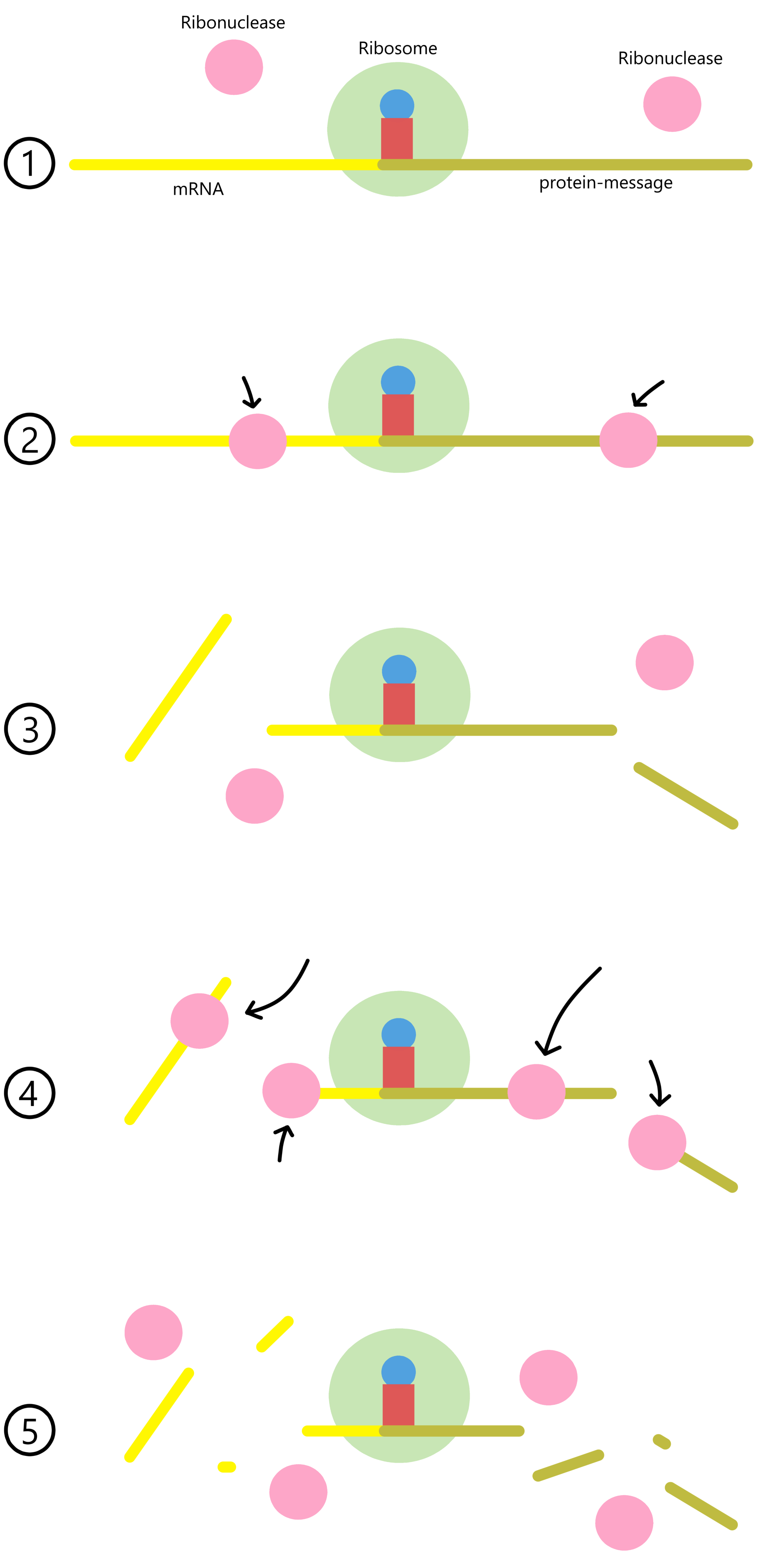
Phage R17 og phage Qβ er virus som kan infisere E. coli-celler og bruke E. coli-ribosomer til å lage virale proteiner. Både R17-RNA og Qβ-RNA inneholder tre protein-meldinger: en A-protein-melding, en Coat-protein-melding, og en Replikase-protein-melding. Initieringsfragmenter fra de tre R17-meldingene ble produsert, sekvensert og publisert i Steitz 1969. På samme tid ble initieringsfragmentet fra Qβ sin Coat-protein-melding sekvensert og publisert i Hindley 1969. Andre initieringsfragmenter ble også publisert senere. Disse sekvensene ble analysert i Shine og Dalgerno's artikkel som diskuteres i neste seksjon.
("Initieringsfragment" er ikke det eneste navnet som brukes på de ribosom-beskyttede basesekvensene. Navnene "initiator fragment", "initiator region", "initiator site", ribosome binding site", og "ribosome attachment site" brukes alle for å referere til ribosom-beskyttede basesekvenser i Steitz 1969 og Hindley 1969. I denne artikkelen vil kun navnet "initiation fragment" benyttes.)
Shine, Dalgarno (1974): The 3'-Terminal Sequence of Escherichia coli 16S Ribosomal RNA: Complementarity to Nonsense Triplets and Ribosome Binding Sites
I 1974 publiserte John Shine og Lynn Dalgarno en artikkel som presenterte basesekvensen i 3'-enden av 16S rRNA (denne rRNA er en av komponentene i ribosomets 30S-del). Deres data, i kombinasjon med data fra andre forskere, viste at sekvensen var 5’-GAUCACCUCCUUA-3’OH. ("OH" indikerer at dette er den faktiske 3'-enden av hele 16S rRNA, ettersom 3'-enden av RNA og DNA har en fri 3'-OH.)
Shine og Dalgarno oppdaget at det i alle de kjente initieringsfragment-sekvensene fantes en kort sekvens som var komplementær til en sekvens i 3'-enden av 16S rRNA. I hvert tilfelle var denne sekvensen lokalisert noen få baser oppstrøms av start-kodonet. Det tydeligste eksempelet ble funnet i initieringsfragment-sekvensen fra phage R17's A-protein-melding, hvor det fantes en syv baser lang sekvens som var komplementær til 3'-enden av 16S rRNA:
|||||||
16S rRNA: 3'OH-AUUCCUCCACUAG-5’
I andre initieringsfragmenter var den komplementære sekvensen noe kortere, noen av den fire baser lange og noen av dem fem baser lange. Shine og Dalgarno tenkte at disse komplementære sekvensene kanskje kunne være steder hvor mRNA og 30S-delen binder sammen under initiering av oversetting, og at sekvensenes baseparing isåfall kanskje gjør at initieringsprosessen går raskere, slik at hver runde med oversetting tar mindre tid og flere proteiner kan produseres.
Steitz, Jakes (1975): How ribosomes select initiator regions in mRNA: Base pair formation between the 3' terminus of 16S rRNA and the mRNA during initiation of protein synthesis in Escherichia coli
Om de komplementære sekvensene i mRNA og 16S rRNA virkelig baseparer sammen under initiering ble testet av Steitz og Jakes in 1975. Sentralt til deres undersøkelse var et protein kalt Colicin E3. E. coli's 16S rRNA er 1541 nukleotider lang, og Colicin E3 kan kutte denne rRNA mellom nukleotidene på posisjon 1492 og 1493. Kuttet produserer to rRNA-fragmenter: et langt fragment (1492 nukleotider langt) som inneholder den opprinnelige 5'-enden, og et kort fragment (49 nukleotider langt) som inneholder den opprinnelige 3'-enden.
Den eksperimentelle metoden kan oppsummeres slik: mRNA-initieringsfragmenter (fra meldingen til phage R17's A-protein) ble blandet med ribosomale 30S- og 50S-deler, initieringsfaktorer (IFs), og initiator-tRNA (i-tRNA). Dette lar komplette 70S-ribosomer settes sammen på mRNA, ved starten av A-protein-meldingen. (mRNA initieringsfragmenter vil heretter refereres til kun som "mRNA".)
Deretter ble Colicin iblandet for å kutte opp 16S rRNA til fragmenter. Dersom initiering av oversetting involverer at 3'-enden av 16S rRNA baseparer sammen med en sekvens i mRNA, så skal det korte 3' rRNA-fragmentet forbli bundet til mRNA, mens det lange 5' rRNA-fragmentet ikke lenger skal være bundet til mRNA.
Sodium dodecyl sulfat (SDS) ble blandet inn for å denaturere de ribosomale proteinene, slik at 70S-ribosomene løses opp i individuelle rRNA og ribosomale proteiner. De oppløste ribosomene ble analysert med gelelektroforese. Dersom mRNA og det korte 3' rRNA-fragmentet er baseparet sammen skal det være mulig å få et gele-bånd som inneholder et kompleks av disse to RNA.
Blanding av mRNA, ribosom-deler, IFs og i-tRNA, samt iblanding av Colicin, ble gjort ved en temperatur på 30°C. Etter at SDS ble blandet inn for å løse opp ribosomene ble blandingen nedkjølt ved å sette den ned i is. Den påfølgende gelelektroforesen ble utført ved en temperatur på 6°C.
Den kalde temperaturen under ribosom-oppløsning og gelelektroforese reduserer RNA-molekylers evne til å basepare sammen. Meningen med eksperimentet er tross alt å teste om mRNA og 3'-enden av 16S rRNA baseparer sammen i løpet av initiering av oversetting, så man må forhindre at mRNA baseparer til annet RNA etter at ribosomene er oppløst. Lavere temperatur gjør også at eksisterende basepar (de som eventuelt har blitt formet under initieringen) blir mer stabile og holder sammen i lengre tid, slik at det blir lettere å analysere dem.)
Resultater: gele-kolonner 1 og 2
Etter gelelektroforese ble RNA i geleen visualisert med to ulike metoder. mRNA hadde blitt produsert med radioaktiv fosfor, så mRNAs posisjon i geleen kunne derfor ses med autoradiografi. Radiografiet av geleen vises i Steitz' Figur 2.
Den andre metoden var bruk at et fargestoff kalt "stains-all" i geleen. Dette fargestoffet binder til både proteiner og RNA, som gjør det mulig å se gele-posisjonen til alle ribosomale komponenter. Steitz viser ikke et vanlig bilde av geleen hvor proteiner og RNA er synlig med stains-all. Istedet brukes tekst overlagt radiografiet for å indikere posisjonen til 5S rRNA, i-tRNA, og de korte 3' rRNA-fragmentene (de korte 3' rRNA-fragmentene refereres til som "Col" for "Colicin fragment". Stedet i radiografiet merket "0" viser start-posisjonen, stedet hvor de ribosomale komponentene ble posisjonert i geleen før elektroforesen.)
Seks ulike prøver ble analysert i geleen. I den første prøven (i kolonne 1) ble mRNA ikke blandet sammen med ribosom-deler eller tRNA eller IFs. Båndet i kolonne 1 viser derfor posisjonen til fritt mRNA (altså mRNA som ikke er bundet til noe annet molekyl).
I den andre prøven (i kolonne 2) ble mRNA blandet med ribosom-deler, tRNA og IFs, før Colicin og deretter SDS ble blandet inn. I kolonne 2 ser vi at båndet som korresponderer til fritt mRNA har blitt svakere, og at et nytt og tregere bånd er tilstede. Dette nye båndet inneholder antakeligvis det baseparede komplekset av mRNA og det korte 3' rRNA-fragmentet.
Resultater: gele-kolonner 3 og 4
Det er mulig at mRNA binder til 16S rRNA i løpet av initiering av oversetting. Men to alternative forklaringer kan også tenkes. Det kan være at en stor andel mRNA ikke binder til ribosomer, og kun binder til de korte rRNA-fragmentene etter at Colinin har kuttet disse fri fra ribosomene. Alternativt kan det være at mRNA binder til ribosomer, men ikke til 16S rRNA, og at binding mellom mRNA og korte 3' rRNA-fragmenter skjer etter at ribosomene har blitt oppløst (til tross for kald temperatur).
To kontroll-prøver ble laget for å utfordre de alternative forklaringene. I den første kontrollprøven ble mRNA, i-tRNA, ribosom-deler og IFs blandet sammen med aurintricarboxylic acid, før Colicin og deretter SDS ble blandet inn. Aurintricarboxylic acid har blitt vist å hindre mRNA i å binde til ribosomet. mRNA i den første prøven vil derfor ha mindre mulighet til å basepare med rRNA i ribosomet.
Dersom mRNA baseparer til 16S rRNA i løpet av initieringen, så kan vi forvente at prøven med aurintricarboxylic acid vil inneholde en mindre mengde baseparet mRNA og korte 3' rRNA-fragmenter. På en annen side, dersom en av de to alternative forklaringene er korrekte, så burde aurintricarboxylic acid ikke utgjøre noen forskjell, og kontrollprøven med acid burde gi samme gele-resultat som prøven i kolonne 2.
Prøven med aurintricarboxylic acid ble puttet i kolonne 3, og som vi kan se så er gelebåndet som korresponderer til baseparet mRNA og rRNA-fragment mindre synlig i kolonne 3 enn i kolonne 2. Dette resultatet går imot de to alternative forklaringene, og støtter tanken om at mRNA og 16S rRNA baseparer i løpet av initiering.
I den andre kontrollprøven ble ribosom-deler, IFs og i-tRNA blandet sammen (uten mRNA), før Colicin og deretter SDS ble iblandet. mRNA ble blandet inn kun etter SDS. Igjen: dersom mRNA og 16S rRNA baseparer i løpet av initiering, så kan vi forvente at denne kontrollprøven vil ha en redusert mengde baseparet mRNA og rRNA-fragmenter. På en annen side, dersom den andre alternative forklaringen er korrekt (mRNA og rRNA-fragmenter baseparer etter at ribosomet er oppløst), så bør den sene iblandingen av mRNA ikke utgjøre noen forskjell.
Den andre kontrollprøven ble puttet i kolonne 4, og som vi kan se så er gelebåndet med mRNA-rRNA-komplekset mindre synlig enn i kolonne 2, noe som går imot den andre alternative forklaringen.
Resultater: mRNA-degradering
En kunne kanskje tro at kolonne 3 og 4 ville hatt omtrent samme mengde fritt mRNA som kolonne 1, men dette er ikke tilfellet. I kolonne 3 er gelebåndet med fritt mRNA veldig svakt sammenlignet med kolonne 1. I kolonne 4, derimot, er båndet med fritt mRNA veldig sterkt, faktisk ser det ut til å være litt sterkere enn i kolonne 1. Det ser derfor ut til at mRNA degraderes til mindre fragmenter, og at det har vært mest degradering i prøven i kolonne 3 og minst degradering i prøven i kolonne 4.
Det er ukjent nøyaktig hva som er årsaken til degraderingen. Kortere mRNA-fragmenter produsert av degradering kan bevege seg raskere gjennom geleen sammenlignet med mRNA i "full størrelse", så de korte fragmentene burde endt opp lenger "ned" i geleen sammenlignet med mRNA i full størrelse. (Om dette er tilfellet eller ikke kan jeg ikke si, fordi Steitz viser ikke et bilde av hele geleen, kun en del av den.)
Det er en detalj ved degraderingen jeg kan kommentere på: det gir mening at degraderingen er minst i den andre kontrollprøven (i kolonne 4), av to årsaker. I denne kontrollprøven iblandes mRNA ganske sent (etter SDS), slik at det som forårsaker degradering har mindre tid til å virke på mRNA. Videre, denne prøven nedkjøles umiddlebart etter at mRNA iblandes, og nedkjølning kan forventes å redusere hastigheten på degraderingsprosessen.
Resultater: gele-kolonner 5 og 6
I den femte prøven ble det undersøkt hva som skjer dersom mRNA blandes med ribosom-deler, i-tRNA og IFs, for deretter å iblandes SDS (mens Colicin ikke benyttes, slik at 16S rRNA ikke kuttes opp i fragmenter). Hvis mRNA binder til 3'-enden av 16S rRNA, og rRNA ikke kuttes opp i korte og lange fragmenter, så skulle mRNA være baseparet til den komplette 16S rRNA.
Den Colicin-frie prøven ble puttet i kolonne 5, og vi ser at størsteparten av mRNA ikke beveger seg fra startstedet i geleen. Dette kan forklares med at den komplette 16S rRNA er for stor til å bevege seg gjennom geleen, slik at rRNA sammen med baseparet mRNA forblir på startstedet. (Steitz skriver at “Fractionation of the reaction mixture on a sucrose gradient (not shown) rather than on a gel directly supports this conclusion; the mRNA fragments co-sediment with the 16S”.)
I kolonne 5 ser vi også at båndet som inneholder mRNA-rRNA-komplekset (det sterke båndet i kolonne 2) er helt fraværende. Dette støtter ideen om at molekylet bundet til mRNA i kolonne 2 faktisk er det korte 3' rRNA-fragmentet, ettersom dette fragmentet ikke eksisterer i den Colicin-frie prøven i kolonne 5.
Kolonne 6 inneholder den samme prøven som kolonne 2 og gir derfor samme resultat som kolonne 2 (Steitz nevner ikke hvorfor denne prøven ble puttet i to kolonner.)
Resultater: gele-kolonne 7
mRNA som benyttes i dette eksperimentet antas å basepare til 3'-enden av 16S rRNA via sekvensene 5’-AGGAGGU-3’ (i mRNA) og 5’-ACCUCCU-3’ (i rRNA). Ifølge en teoretisk beregning burde en temperatur på 55°C være varmt nok til å “smelte” (bryte opp) baseparene mellom de to sekvensene.
I den siste prøven ble mRNA, ribosom-deler, IFs og i-tRNA blandet sammen, Colicin ble iblandet, og ribosomene ble oppløst med SDS. De oppløste ribosomene ble så varmet til 55°C i fem minutter før de ble puttet i kolonne 7 i geleen.
Dersom mRNA og rRNA kun holdes sammen av basepar mellom de to nevnte sekvensene, så burde varmebehandlingen gjøre at baseparede mRNA og rRNA-fragmenter separeres til frie mRNA og frie rRNA-fragmenter (forutsatt at den teoretiske beregningen er riktig). Hvis varmebehandlingen ikke forårsaker separering av mRNA og rRNA-fragmenter ville dette altså antyde at de to holdes sammen på et annet vis enn med basepar, for eksempel med et kovalent bånd.
I kolonne 7 ser vi at det ikke er noe synlig bånd på posisjonen som korresponderer til mRNA-rRNA-komplekset, mens båndet for fritt mRNA en noe sterkere enn i kolonner 2 og 6. Dette bekrefter at mRNA-rRNA-komplekset smelter under varmebehandlingen. Dette resultatet beviser ikke at mRNA og rRNA holdes sammen av basepar mellom AGGAGGU og ACCUCCU, men det viser ihvertfall at det ikke er noen kovalent binding mellom mRNA og rRNA.
Kommentar
I kolonne 5 blir størsteparten av mRNA værende på startposisjonen i geleen, noe som kan forklares med at mRNA baseparer til komplette 16S rRNA som er for store til å bevege seg gjennom geleen. Men vi ser også at noe mRNA blir værende på startposisjonen i alle de andre kolonnene unntatt kolonne 1 (hvor prøven kun inneholder fritt mRNA).
Jeg er ikke sikker på hvorfor dette skjer. I tilfellene med kolonne 2 og 6 kan en tenke at Colicin kanskje ikke kuttet alle 16S rRNA, slik at en liten mengde rRNA fremdeles er komplette og kan holde igjen en liten mengde mRNA på startposisjonen. En slik forklaring kan dog ikke brukes for kolonne 3, 4 eller 7. I prøvene i kolonne 3 og 4 så binder ikke mRNA til 16S rRNA, enten fordi aurintricarboxylic acid hindrer mRNA i å binde ribosomet, eller fordi RNA iblandes kun etter at ribosomene er oppløst med SDS. Og i prøven i kolonne 7 vil varmebehandlingen få baseparet mRNA og rRNA til å smelte og bli til frie mRNA og frie rRNA-fragmenter. Det ser derfor ut til at det er noe annet enn 16S rRNA som holder igjen litt av mRNA på startposisjonene.
Alt i alt så støtter Steitz' resultat hypotesen om at en sekvens i mRNA oppstrøms av et start-kodon kan basepare til 3'-enden av 16S rRNA i løpet av initiering av oversetting.
SD- and aSD-sequences
På et tidspunkt (jeg tror sent i 1970-årene eller tidlig i 1980-årene) ble det generelt akseptert at mRNA og 3'-enden av 16S rRNA baseparer sammen i løpet av initiering. Når dette skjedde begynte mRNA-sekvenser lokalisert like oppstrøms av start-kodoner og komplementære til 3'-enden av 16S rRNA å bli referert til som “Shine-Dalgarno-sekvenser” (SD-sekvenser), mens en sekvens i 3'-enden av 16S rRNA som kan binde til en SD-sekvens begynte å bli referert til som “anti-Shine-Dalgarno-sekvenser” (aSD-sekvenser).
For eksempel, hos meldingen for phage R17 A-protein så er SD-sekvensen 5'-AGGAGGU-3' og aSD-sekvensen er 5'-ACCUCCU-3':
|||||||
rRNA: 3'OH-AUUCCUCCACUAG-5’
Andre protein-meldinger kan ha andre SD-sekvenser som binder til andre aSD-sekvenser.
(Som nevnt i seksjonen "Initieringsfragmenter", navnet "ribosome binding site" er et alternativt navn for "initiation fragment". Til tross for dette så er det mange tekster, inkludert biologi-lærebøker, som benytter "ribosome binding site" som et alternativt navn for SD-sekvensen. Denne bruken av det samme navnet for forskjellige ting kan skape unødvendig forvirring, så jeg synes at "ribosome binding site" derfor ikke bør brukes som et alternativt navn på SD-sekvensen.)
Stormo, Schneider, Gold (1982): Characterization of translational initiation sites in E. coli
Innen 1982 hadde sekvensen rundt start-kodonet i mange E. coli protein-meldinger blitt bestemt. Disse sekvensene er samlet i Stormo 1982. I alle unntatt en av meldingene inneholdt de femten basene oppstrøms av start-kodonet en sekvens, minst tre baser lang, som var komplementær til 3'-enden av 16S rRNA. Et utvalg av disse sekvensene vises her:
araC: 5’-GGGAGUAUGAAAAGUAUG-3’ (lengde=4, spacer=10)
pheA: 5’-CAAAAAGGCAACACUAUG-3’ (lengde=3, spacer=7)
recA: 5’-AUGACAGGAGUAAAAAUG-3’ (lengde=5, spacer=5)
rpsD(S11): 5’-CGGGGUGAUUGAAUAAUG-3’ (lengde=6, spacer=6)
trpR: 5’-CAAUGGCGACAUAUUAUG-3’ (ingen SD-sekvens)
Som Schurr 1993 har bemerket, SD- og aSD-sekvenser trenger ikke nødvendigvis å være kontinuerlige. I hvertfall i teorien så kan baseparede SD-aSD-sekvenser inneholde "bulged nucleotides" eller "looped nucleotides". Et eksempel på en bulged nucleotide vises i Schurr's Figur 1. En kan se at hos rpsD så kan en bulge i aSD-sekvensen tillate at SD-sekvensen forlenges til 5'-GGGGUGAU-3'. Denne komplikasjonen med bulges og loops vil ignoreres i denne artikkelen, da det ikke er viktig her.
Hui, de Boer (1987): Specialized ribosome system: Preferential translation of a single mRNA species by a subpopulation of mutated ribosomes in Escherichia coli
(Mens binding mellom SD- og aSD-sekvensene støttes av resultatet til Steitz 1975, så har jeg ikke så langt vist at bindingen bidrar til at initieringsprosessen går raskere. La oss for nå bare anta at dette er sant. Resultatet presentert i denne seksjonen vil støtte antakelsen.)
3'-enden av 16S rRNA har sekvensen 5'-GAUCACCUCCUUA-3’OH. Mange mRNA har SD-sekvenser som kan binde til minst tre av basene 5'-CCUCC-3' i 16S rRNA. For eksempel så kan SD-sekvensen oppstrøms av lacZ-meldingen, AGGA, binde til aSD-sekvensen UCCU, som involverer tre av de fem CCUCC-basene:
||||
16S rRNA: 3'OH-AUUCCUCCACUAG-5’
Derfor kan mutasjoner i CCUCC-sekvensen i 16S rRNA som forhindrer dens baseparing til SD-sekvenser forventes å redusere hastigheten på initiering-prosessen for mange mRNA.
Hva om CCUCC-sekvensen i 16S rRNA ble mutert så den ikke kan binde til SD-sekvensen, og en komplementær mutasjon ble introdusert i SD-sekvensen til en protein-melding, slik at SD-sekvensens evne til å basepare med aSD-sekvensen gjenopprettes? Ville dette gjenopprette effektiv initiering for denne spesifikke meldingen? Dette er det som Hui og de Boer forsøkte å gjøre: lage E. coli-celler med mutant-ribosomer som oversetter en bestemt mutert protein-melding mer effektivt enn andre meldinger i cellene.
Metode
Kromosomalt DNA i E. coli inneholder syv gener for 16S rRNA. Disse genene er rrsA, rrsB, rrsC, rrsD, rrsE, rrsG, rrsH. Et plasmid med en kopi av rrsB ble satt inn i en kultur av E. coli-celler, slik at cellene fikk totalt åtte gener for 16S rRNA.
Den innsatte plasmiden innneholdt også et hGH-gen med SD-sekvensen GGAGG. hGH-meldingen vil fungere som den spesifikke meldingen nevnt ovenfor. Plasmidet inneholdt altså et hGH-gen med SD-sekvensen GGAGG og et rrsB-gen med aSD-sekvensen CCUCC. Dette vil refereres til som "referanse-plasmidet".
To andre plasmider ble også laget. I det ene var hGH sin SD-sekvens mutert til CCUCC, mens rrsB sin aSD-sekvens var mutert til GGAGG. Dette vil refereres til som det "inverterte plasmidet". I et annet plasmid var SD-sekvensen mutert til GTGTG, mens aSD-sekvensen var mutert til CACAC. Dette vil refereres til som det "alternative plasmidet". De to plasmidene ble satt inn i to E. coli-kulturer.
I alle tre plasmidene ble transkripsjon av rrsB-genet kontrollert av en lambda PL operator-sekvens sammen med et lambda cI857 repressor-protein. cI857-repressoren er aktiv ved en temperatur på 30°C og binder da til PL-operatoren, slik at transkripsjon av rrsB inhiberes. Ved høyere temperaturer, slik som 38°C, så kan ikke cI857-repressoren opprettholde sin aktive struktur og kan ikke binde til PL-operatoren. Plasmidets rrsB-gen vil derfor transkripteres ved 38°C. De syv kromosomale genene for 16S rRNA blir transkriptert uavhengig av temperatur.
Dette betyr at ved 30°C vil alle ribosomene inneholde 16S rRNA fra de syv kromosomale genene, med den u-muterte aSD-sekvensen CCUCC. Ved 38°C vil noen ribosomer inneholde 16S rRNA fra de kromosomale genene, med u-mutert aSD CCUCC, mens noen ribosomer vil inneholde 16S rRNA fra rrsB-genet i plasmiden, med aSD-sekvens CCUCC eller GGAGG eller CACAC (avhengig av plasmiden).
(Plasmidenes hGH-gen var ikke kontrollert av noen repressor og ble transkriptert hele tiden uavhengig av temperatur. Det var derfor forventet av konsentrasjonen av hGH-mRNA ville være rimelig konstant, og at en endring i produksjon av hGH-protein når temperaturen øker ikke kan være forårsaket av en endring i konsentrasjonen av hGH-mRNA.)
De tre E. coli-kulturene ble først dyrket på 30°C, før temperaturen ble økt til 38°C. Oversettingen av hGH-meldingen ble bestemt indirekte ved å måle konsentrasjonen av hGH-proteiner i hver av de tre kulturene. (hGH-protein er "human growth hormone", et protein som ikke finnes naturlig i E. coli. All hGH-protein som finnes i cellene stammer derfor fra oversetting av hGH-genet i plasmiden.)
Resultater
Konsentrasjonen av hGH-protein vises i Hui og Boer's Figur 3, hvor tidspunkt 0 er øyeblikket da temperaturen ble økt fra 30°C til 38°C.
Målinger fra kulturen med referanse-plasmidet, representert i figuren med △, viser at oversetting av hGH-meldingen i denne kulturen er relativt høy ved 30°C og forblir høy ved 38°C (altså: hGH-meldingen blir oversatt effektivt uavhengig av temperaturen). Dette var som forventet, ettersom en melding med SD-sekvensen GGAGG kan oversettes av ribosomer som inneholder 16S rRNA fra de kromosomale genene (som transkripteres uavhengig av temperatur).
Målinger fra kulturen med den inverterte plasmiden, representert med ◆, og fra kulturen med den alternative plasmiden, representert med ●, viser at hGH-meldinger med de muterte SD-sekvensene CCUCC og GTGTG oversettes svært ineffektivt ved 30°C. Dette er som forventet, da meldinger med slike SD-sekvenser ikke kan binde til aSD-sekvensen i 16S rRNA fra de kromosomale genene, og ved 30°C vil alle ribosomene ha slike 16S rRNA.
Etter at temperaturen har blitt økt til 38°C i kulturene med inverterte og alternativt plasmider, så vil rrsB-genet i plasmidene transkripteres, og en del av de nye ribosomene som settes sammen vil inneholde aSD-sekvensen GGAGG eller CACAC. Vi ser at ved denne temperaturen blir oversetting av hGH-meldingen mer effektiv, da det er en gradvis akkumulering av hGH-protein. Dette resultatet støtter tanken om at SD-aSD-baseparing gjør at initieringsprosessen går raskere, slik at oversetting generelt går raskere.
Et uventet resultat er at 3 timer etter at temperaturen ble økt så er konsentrasjonen av hGH-protein omtrent dobbelt så stor i kulturen med det alternative plasmidet, sammenlignet med kulturen med det inverterte plasmidet. Dette antyder at SD- og aSD-sekvensene i det alternative plasmidet (SD:GTGTG/aSD:CACAC) av en eller annen grunn fungerer bedre enn sekvensene i den inverterte plasmiden (SD:CCTCC/aSD:GGAGG).
Kontroll-prøver
For kulturene med den inverterte plasmiden og den alternative plasmiden, så tenkte Hui og Boer at det var en mulighet for at den økte konsentrasjonen av hGH-protein ved 38°C ikke var relatert til transkripsjon av plasmidets rrsB-gen. De tenkte at økningen i temperatur fra 30°C til 38°C muligens kunne forårsake en eller annen uforutsett endring i E. coli-cellene, og at dette kunne være årsaken til økningen i mengden hGH-protein. For å teste denne muligheten ble to kontroll-plasmider laget.
En kontroll-plasmid inneholdt et hGH-gen med den muterte SD-sekvensen CCTCC (samme SD som den inverterte plasmiden), mens en annen kontroll-plasmid inneholdt et hGH-gen med SD-sekvensen CACAC (samme SD som det alternative plasmidet). Forskjellen mellom de to kontroll-plasmidene og det inverterte/alternative plasmidet var at kontroll-plasmidene ikke inneholdt et rrsB-gen.
Dersom en økning av hGH-protein ved 38°C ikke var forårsaket av transkripsjon av plasmidens rrsB-gen, så vil fjerning av rrsB-genet fra plasmidet ikke ha noen påvirkning på mengden hGH-protein som produseres. Isåfall burde de to kontroll-kulturene med hver sin kontroll-plasmid ha samme produksjon av hGH-protein som kulturene med det inverterte/altenative plasmidet.
Målinger av hGH-protein i de to kontroll-kulturene, representert med □ og ○, viser at temperaturøkning fra 30°C til 38°C ikke skaper økt oversetting av hGH-meldinger med muterte SD-sekvenser. Oversetting av hGH-meldinger med muterte SD-sekvenser må derfor være avhengig av ribosomer med muterte aSD-sekvenser (altså: avhengig av transkripsjon av plasmidets rrsB-gen).
(Jeg tenker kontroll-plasmidene ideelt sett burde ha inneholdt u-muterte rrsB-gener med aSD-sekvens CCTCC, for å sikre at ulike SD-sekvenser hos hGH-meldingen er den eneste variabelen som skiller kontroll-plasmidene fra referanse-plasmidet.)
Jacob, Santer, Dahlberg (1987): A single base change in the Shine-Dalgarno region of 16S rRNA of Escherichia coli affects translation of many proteins
(Jeg synes egentlig at resultatene fra Jacob 1987 ikke er så veldig interessante, og de inkluderes her hovedsakelig på grunn av Jacob's bruk av to-dimensjonal gelelektroforese. Jeg har tenkt å skrive en artikkel om gelelektroforese snart, og likte derfor at denne artikkelen gir et praktisk eksempel på bruk av 2D-gelelektroforese.)
Som vi så i forrige seksjon så testet Hui og Boer hvordan en fem-basers mutasjon i 16S rRNA påvirket oversettingen av en enkelt protein-melding, nemlig hGH-meldingen. Jacob utførte et noenlunde lignende eksperiment, hvor han testet hvordan en basemutasjon i E. colis 16S rRNA påvirker oversettingen av mange forskjellige protein-meldinger. I u-muterte 16S rRNA er sekvensen ved 3'-enden:
Mens i mutanten brukt i Jacob's eksperiment er sekvensen:
Det muterte rrsB-genet ble satt inn i en plasmid. Transkripsjon av rrsB-genet ble kontrollert av lambda PL operator-sekvensen sammen med lambda cI857 repressor-proteinet. Plasmiden ble satt inn i en kultur av E. coli-celler. En kontroll-kultur med et plasmid med et u-mutert rrsB-gen ble også laget.
Mens de to kulturene vokste på 42°C for å tillate transkripsjon av plasmidenes rrsB-gener, så ble radioaktiv [35S]methionin blandet inn i kulturene for å gjøre nylig produserte proteiner radioaktive. Cellene hadde 20 minutter til å inkorporere [35S]methionin i proteiner før alle proteinene ble ekstrahert fra begge kulturene. De to protein-ekstraktene ble analysert med todimensjonal gelelektroforese (hvor proteinene først separeres i en dimensjon via isoelektrisk fokusering, og deretter separeres i en annen dimensjon ut i fra protein-masse).
Resultater
Geleene vises i Jacob's figur 4, hvor 4a er geleen med proteiner fra kontroll-kulturen og 4b er geleen med proteiner fra mutant-kulturen.
Jacob's analyse av protein-mønstrene fokuserer på noen få protein-flekker som oppfyller to kriterier: først, flekken må bare inneholde én enkelt type protein, og det proteinet må være kjent (slik informasjon kommer fra tidligere arbeid av andre forskere). For eksempel, protein-flekken markert med "A" inneholder et protein kalt “alpha subunit of (Ca2+,Mg2+)-ATPase” (kort navn: AtpA). Og for det andre, SD-sekvensen til protein-meldingen må være kjent. Ifølge Jacob's tabell 1 så er SD-sekvensen for AtpA:
(To mulige SD-sekvenser vises med fet tekst hvor enten av sekvensene muligens er funksjonelle. Start-kodonet er understreket. Dersom en bulging nukleotid tillates i aSD-sekvensen så kan SD-sekvensen 5'-AGGGG-3' muligens også være funksjonell.)
På den tiden eksperimentet ble utført var det kun AtpA og syv andre proteiner som oppfylte begge kriteriene. Disse er listet i Jacob's tabell 1. (Av en eller annen grunn er det kun fem av de åtte proteinene som er indikert på geleen i Jacob's figur 4. I min kommentar på resultatet vil jeg ignorere de tre proteinene som ikke er merket på geleen.)
Som nevnt, flekken som er markert med "A" inneholder proteinet AtpA. Denne flekken er mindre (inneholder mindre protein) i figur 4b enn i 4a, noe som antyder at 16S rRNA-mutasjonen fører til redusert oversetting av dette proteinet. AtpA-meldingens SD-sekvenser kan basepare med 16S rRNA på følgende måter:
|||
rRNA: 3’OH-AUUCCUCCACUAG-5’
eller alternativt:
||||
rRNA: 3’OH-AUUCCUCCACUAG-5’
Med den muterte 16S rRNA reduseres antall basepar som kan dannes mellom de to sekvensene:
| |
rRNA: 3’OH-AUUCUUCCACUAG-5’
eller alternativt:
| ||
rRNA: 3’OH-AUUCUUCCACUAG-5’
Den reduserte mengden AtpA-protein i kulturen med den muterte 16S rRNA korrelerer derfor med en svakere baseparing mellom SD- og aSD-sekvensene.
Protein-flekken markert "D" inneholder proteinet AtpB ("beta subunit of (Ca2+,Mg2+)-ATPase"). Som med AtpA så viser AtpB er korrelasjon mellom redusert flekk-størrelse i figur 4b og svakere baseparing mellom SD-sekvensen og den muterte aSD-sekvensen.
Flekken markert "B" inneholder proteinet MetRS (methionyl-tRNA synthetase), og i dette tilfellet er flekken større (inneholder mer protein) i figur 4b enn i 4a, noe som antyder at 16S rRNA-mutasjonen fører til økt oversetting av dette proteinet. MetRS-meldingens SD-sekvens kan basepare med 16S rRNA på denne måten:
|||| ||
rRNA: 3’OH-AUUCCUCCACUAG-5’
Med den muterte 16S rRNA økes antall basepar som kan dannes mellom de to sekvensene:
|||||||
rRNA: 3’OH-AUUCUUCCACUAG-5’
Den økte mengden MetRS i kulturen med den muterte 16S rRNA korrelerer altså med en sterkere baseparing mellom SD- og aSD-sekvensene.
(Flekken markert "C" inneholder ribosomalt protein S1. I figur 4a har denne flekken en lang "hale" på venstre side, mens i 4b er det ikke noen slik hale. Jeg synes at den relative mengde protein i de to flekkene ikke kan bestemmes visuelt når flekkene har forskjellig form, so jeg vil ikke kommentere på "C"-flekken.)
Flekken markert "E" inneholder MetK (S-adenosyl-methionine synthetase). Denne flekken har en ganske lik størrelse (lik mengde protein) i både figur 4a og 4b, noe som antyder at mutasjonen i 16S rRNA ikke påvirker oversettingen. MetK-meldingens SD-sekvens kan basepare med 16S rRNA på denne måten:
||||||
rRNA: 3’OH-AUUCCUCCACUAG-5’
Med den muterte 16S rRNA forblir antall basepar uendret:
||||||
rRNA: 3’OH-AUUCUUCCACUAG-5’
Den uforandrede mengden MetK i kulturen med den muterte 16S rRNA korrelerer derfor med en uforandret baseparing mellom SD- og aSD-sekvensene.
Selv om Jacob's resultater er kompatible med hypotesen til Shine og Dalgarno, om at baseparing mellom SD- og aSD-sekvensene påvirker hastigheten til initiering av oversetting, så tenker jeg at arbeidet holdes tilbake av at de kun kan analysere noen få proteiner. Det hadde vært interessant å se om korrelasjonen mellom mengden protein og SD-aSD-baseparing ble opprettholdt i et større sett med proteiner.
Chen, Bjerknes, Kumar, Jay (1994): Determination of the optimal aligned spacing between the Shine-Dalgarno sequence and the translation initiation codon of Escherichia coli mRNAs
Ulike protein-meldinger har ulik avstand ("spacing") mellom SD-sekvensen og start-kodonet. For eksempel, E. coli’s recA-melding har en "spacer" på fem nukleotider mellom SD-sekvensen og start-kodonet, rpsD-meldingen har en spacer på seks nukleotider, lacZ-meldingen har en spacer på syv nukleotider, og araC-meldingen har en spacer på ti nukleotider.
rpsD: 5’-CGGGGUGAUUGAAUAAUG-3’ (spacer:6)
lacZ: 5’-ACACAGGAAACAGCUAUG-3’ (spacer:7)
araC: 5’-GGGAGUAUGAAAAGUAUG-3’ (spacer:10)
Chen tenkte at baseparing mellom SD-sekvensen og anti-SD-sekvensen hjelper med å posisjonere start-kodonet ovenfor antikodonet på initiator-tRNA i ribosomets P-sted, for å promotere bindingen mellom kodon og antikodon som må ta sted i løpet av initieringsprosessen. Hvis dette stemmer, så burde det finnes en optimal lengde av "spaceren" mellom SD-sekvensen og start-kodonet, en lengde som tilsvarer avstanden mellom aSD-sekvensen og antikodonet/P-stedet. Chen ønsket å finne denne optimale spacer-lengden.
Søket etter den optimale spaceren kompliseres ved at ulike SD-sekvenser binder ulike aSD-sekvenser med ulik avstand fra antikodonet/P-stedet. For eksempel, la oss for en stund bare anta at det er en syv basers distanse mellom P-stedet og aSD-sekvensen UCCU (jeg sier ikke at dette er sant, antakelsen er kun for demonstrasjons skyld). Hvis dette er tilfellet, så vil lacZ-meldingens start-kodon være perfekt posisjonert ovenfor P-stedet:
tRNAfMetUAC
16S rRNA: 3'OH-AUUCCUCCA-5' UAC
|||| |||
lacZ: 5’-ACACAGGAAACAGCUAUG-3’
Se nå på pheA-meldingen. Som med lacZ-meldingen så har pheA-meldingen en 7-base spacer mellom SD-sekvensen og start-kodonet:
Men når pheA-meldingens SD-sekvens baseparer med 16S rRNA så vil start-kodonet ikke være perfekt posisjonert ovenfor P-stedet slik som var tilfellet med lacZ-meldingen, selv om begge meldingene har en 7-base spacer:
tRNAfMetUAC
16S rRNA: 3'OH-AUUCCUCCA-5' UAC
|||
pheA: 5’-CAAAAAGGCAACACUAUG-3’
Dette betyr at selv om en klarte å finne den optimale lengden av spaceren for en bestemt SD-sekvens, så skulle andre SD-sekvenser ha andre optimale spacere.
For å generalisere om den optimale lengden av spaceren er det derfor nødvendig å benytte konseptet "aligned spacing". Basen A1534 velges som et vilkårlig referansepunkt (dette er en Adenin som er base #1534 når man teller fra 5'-enden av 16S rRNA, eller base #9 når man teller fra 3'-enden). Aligned spacing refererer til det antallet baser som skiller en meldings start-kodon fra den basen som "aligner" med A1534. For eksempel så har lacZ-meldingen en aligned spacing på fire baser:
16S rRNA: 3'OH-AUUCCUCCA-5' UAC
|||| : |||
lacZ: 5’-ACACAGGAAACAGCUAUG-3’ (aligned spacing: 4)
Mens pheA-meldingen har en aligned spacing på tre baser:
16S rRNA: 3'OH-AUUCCUCCA-5' UAC
||| :
pheA: 5’-CAAAAAGGCAACACUAUG-3’ (aligned spacing: 3)
Dersom funksjonen til SD-aSD-baseparingen virkelig er å posisjonere start-kodonet ovenfor antikodonet/P-stedet, så burde det finnes en optimal aligned spacing som er universell for alle SD-sekvenser. Dette fordi avstanden mellom A1534 og antikodonet/P-stedet er uavhengig av SD-sekvensen.
(I figurene ovenfor ser det ut som at start-kodonet i pheA-meldingen ikke kan posisjoneres ovenfor P-stedet, men dette er ikke virkelig tilfellet. mRNA og ribosomet er ikke helt rigide strukturer, de har litt fleksibilitet. Dette gjør det mulig for start-kodonet å posisjoneres ovenfor P-stedet selv med suboptimale spacere, men posisjoneringen vil da ta mer tid.)
Metode
Et protein-melding med en optimal lengde på spaceren burde ha en høyere grad av oversetting enn samme melding med en annen lengde på spaceren. Chen valgte å bruke cat-meldingen for å teste ulike spacere. Oversettelse av cat-meldingen kan måles indirekte ved å måle aktiviteten til CAT-proteinet (chloramphenicol acetyltransferase).
Chen brukte oligonukleotid-syntese til å produsere to serier med korte DNA: d-serien inneholdt SD-sekvensen TAAGG samt start-kodonet AUG, og ble laget med elleve ulike spacer-lengder, mens D-serien inneholdt SD-sekvensen GAGGT samt AUG, og ble laget med ni ulike spacer-lengder (se Chen’s Figur 3). Hver av de tyve korte DNA ble brukt til å erstatte SD-sekvensen og start-kodonet til et cat-gen i en plasmid, og de resulterende tyve plasmidene ble transformert inn i hver sin E. coli-kultur.
Resultater
I hver kultur ble aktiviteten til CAT-proteinet undersøkt (se Chen’s Figur 4). For SD-sekvensen GAGGT var aktiviteten høyest når spaceren var fem baser lang, og aktiviteten faller gradvis av med spacere som er lengre eller kortere enn fem baser. For SD-sekvensen TAAGG var CAT-aktiviteten høyest når spaceren var ni baser lang, og igjen faller aktiviteten gradvis av med spacere som er lengre eller kortere enn ni baser. I begge SD-seriene var CAT-aktiviteten høyest med en aligned spacing på fem baser. Dette resultatet støtter hypotesen om at funksjonen til SD-aSD-baseparingen er å posisjonere start-kodonet ovenfor antikodonet på initiator-tRNA i ribosomets P-sted.
Det nevnes to svakheter ved eksperimentet. Det ene er at konsentrasjonen til CAT-mRNA ikke måles. Det er derfor mulig at ulik lengde på spaceren påvirker raten av transkripsjon og/eller mRNA-degradering, noe som vil påvirke CAT-aktiviteten.
Det andre er at lengden på sekvensen 5’ for start-kodonet, altså lengden på ledesekvensen, kan påvirke raten av oversetting. Mens lengden på spaceren varierer mellom 2 og 17 nukleotider så varierer lengden på ledesekvensen mellom 35 og 50 nukleotider. Ideelt sett burde ledesekvensen ha konstant lengde uavhengig av spaceren. I praksis betyr dette at meldinger med færre nukleotider i spaceren burde hatt flere nukleotider 5’ for SD-sekvensen.
Det kommenteres også på at kultur-serien med D-serien av plasmider (SD:GAGGT) har en maksimal CAT-aktivitet på 0.9 enheter, mens kultur-serien med d-serien av plasmider (SD:TAAGG) kun har en maksimal aktivitet på 0.4 enheter. Det er ikke sikkert hva som er årsaken til denne forskjellen.
Vimberg, Tats, Remm, Tenson (2007): Translation initiation region sequence preferences in Escherichia coli
For å finne ut hvordan lengden av SD-sekvensen påvirker produksjonen av proteiner ble SD-sekvensens lengde variert fra 1 base til 8 baser (se Vimberg's figur 1). Hver SD-sekvens ble plassert foran et gen for GFP (green fluorescent protein) i en plasmid som ble satt inn i en E. coli-kultur. Konsentrasjonen av GFP i hver kultur ble bestemt ved å måle kulturens fluorescens, mens konsentrasjonen av GFP-mRNA ble bestemt med kvantitativ polymerase chain reaction. Effektiviteten av oversettingen av GFP presenteres som GFP's "expression level", altså mengden GFP produsert per mRNA.
Når E. coli ble dyrket ved en temperatur på 37°C var det den 6 baser lange SD-sekvensen 5'-AGGAGG-3' som ga høyest expression (se Vimberg's figur 2). Den 1 base lange "SD-sekvensen" med én enkelt G-base ga som forventet lavest expression. Hver lengdeøkning fra 1 til 2 til 3 til 4 til 5 til 6 forårsaket en økning i expression. Ytterlige lengdeøkninger fra 6 til 7 og fra 7 til 8 forårsaket en reduksjon i expression.
Effekten av SD-lengden ble funnet å være avhengig av fravær eller tilstedeværelse av en såkalt "enhancer-sekvens" oppstrøms av SD-sekvensen. I fravær av en enhancer var den 6 baser lange SD omtrent 1.5 til 2.5 ganger mer effektiv enn den 1 base lange SD. Når en enhancer var tilstede var den 6 baser lange SD ti ganger mer effektiv enn den 1 base lange SD.
Ettersom lengden av SD-sekvensen øker blir baseparingen mellom SD- og aSD-sekvensene mer stabil, slik at start-kodonet får bedre hjelp i å posisjonere seg ovenfor ribosomets P-sted. Det kan tenkes at dersom SD-sekvensen er "for lang" og SD-aSD-bindingen er "for stabil", så blir det vanskelig for ribosomet å bryte seg fri fra SD-sekvensen for å begynne å bevege seg langs mRNA og går over fra initiering til elongering. Dette kan forklare reduksjonen i expression som vi ser i SD-sekvensene med lengde 7 og 8 sammenlignet med SD-sekvensen med lengde 6.
E. coli-celler ble også dyrket på 20°C, og da var det den fem baser langs SD-sekvensen GGAGG som ga høyest oversettelse (se Vimberg’s Figur 3). Det er ikke bare økt lengde på SD-sekvensen som gir økt stabilitet av SD-aSD-bindingen, lavere temperatur fører også til økt stabilitet av bindingen. Resultatet på 20°C støtter derfor tanken om at en "for stabil" SD-aSD-binding reduserer oversettelsen i de lengste SD-variantene.
Saito, Green, Buskirk (2020): Translational initiation in E. coli occurs at the correct sites genome-wide in the absence of mRNA-rRNA base-pairing
Så langt i denne artikkelen har vi sett resultater som gir god støtte til hypotesen om at SD- og aSD-sekvensene binder sammen i løpet av initiering av oversetting, og at binding-stabiliteten og posisjonen til de to sekvensene kan påvirke hvor effektivt proteiner produseres. Noen betydelige spørsmål har ikke blitt besvart ennå: blant de omtrent 4300 genene i E. coli, hvor mange gener har SD-sekvenser? Og for de genene som har SD-sekvenser, har bindingsstabilitet og posisjonen av SD- og aSD-sekvensene god korrelasjon med hvor effektivt disse genene oversettes til proteiner? I denne siste seksjonen skal vi se svaret på ett av disse spørsmålene.
Saito brukte "ribosome occupancy" som et mål for oversettings-effektivitet i et stort sett protein-meldinger fra E. coli. En melding's ribosome occupancy refererer til hvor nært sammen ribosomene befinner seg på meldingen (husk at en melding kan oversettes av flere ribosomer samtidig). En kortere distanse mellom ribosomene betyr høyere ribosome occupancy, noe som indikerer mer effektiv oversettelse.
For hver melding ble "styrken" på meldingens SD-sekvens beregnet i form av −ΔG. (ΔG, endring i fri energi, representerer her den termodynamiske stabiliteten til de baseparede SD-aSD-sekvensene, hvor lavere (mer negative) verdier av ΔG indikerer høyere stabilitet. Derfor vil høyere (mer positive) verdier av −ΔG indikere høyere stabilitet.) I hele det analyserte settet med meldinger ble bare en svak korrelasjon (0.11) funnet mellom ribosome occupancy og SD-styrke (se Saito's figur 2A).
Meldingenes ribosome occupancy ble også testet i en annen E. coli-kultur hvor ribosomene inneholdt 16S rRNA med mutasjoner i 3'-enden. CCUCC-sekvensen i 3'-endene hadde blitt endret til AAAAA, slik at de muterte ribosomene ikke kunne binde meldingenes SD-sekvenser. For denne kulturen ble det funnet en sterk negative korrelasjon (-0.50) mellom ribosome occupancy og SD-styrke. (se Saito's figur 2B. Merk at SD-styrke i figur 2B representerer stabiliteten til baseparingen mellom SD-sekvensen og den u-muterte aSD-sekvensen, ikke mellom SD-sekvensen og den muterte aSD-sekvensen.)
Med andre ord, i den andre kulturen med muterte ribosomer som ikke binder til SD-sekvenser, så blir meldinger med svakere SD-sekvenser oversatt mer effektivt enn meldinger med sterke SD-sekvenser. En mulig forklaring på dette resultatet er at sterke SD-sekvenser korrelerer med en eller flere andre faktorer som gjør oversettingen mindre effektiv.
For hver melding i settet beregnet Saito forskjellen i ribosome occupancy mellom kulturen med vanlige ribosomer og kulturen med muterte ribosomer. Forskjellen i ribosome occupancy hadde en sterk positiv korrelasjon (0.68) med SD-styrke (se Saito's figur 2C). Dette betyr at generelt sett, desto sterkere SD-sekvens en melding har, desto mer effektivt kan den oversettes av ribosomer som kan binde til SD-sekvensen, i forhold til ribosomer som ikke kan binde til SD.
Kort oppsummert så viser resultatene fra figur 2A, 2B og 2C at selv om SD-sekvenser bidrar til mer effektiv oversetting av meldinger i E. coli, så blir styrken på SD-sekvensen generelt ikke brukt for å justere graden av oversetting av individuelle meldinger (fordi sterke SD-sekvensen pleier å korrelere med faktorer som skaper redusert oversetting).
Kilder
Hindley, Staples
(1969): Sequence of a Ribosome Binding Site in Bacteriophage Qβ-RNA. Betalt artikkel
Schurr, Nadir, Margalit
(1993): Identification and characterization of E.coli ribosomal binding sites by free energy computation. Fri artikkel
Steitz
(1969): Polypeptide chain initiation: nucleotide sequences of the three ribosomal binding sites in bacteriophage R17 RNA. Betalt artikkel